 |
 |
[Entrada] [Actividades]
[Revista I + S] [Solicitud de
Inscripción]
BIOINFORMÁTICA Y SALUD:
Impactos de la aplicación de las nuevas tecnologías para el tratamiento
de la información genética
en la investigación biomédica y la práctica clínica
Fernando Martín Sánchez; Guillermo López Campos; Víctor Maojo García (*)
Unidad de Bioinformática - BIOTIC
Subdirección General de Laboratorios y Servicios en Salud Pública
Instituto de Salud Carlos III
(*) Grupo de Informática Médica – Facultad de Informática – U.P.M.
"Averiguar la secuencia de nuestros genomas personales podrá ser algún día una actividad tan común como es hoy obtener el grupo sanguíneo..."
Andrei Mirzabekov- Argonne Nat. Lab. (JAMA Medical News & Perspectives - Nov 20, 1996)
"Las nuevas tecnologías basadas en biochips, junto con la información del genoma humano, ayudarán a los médicos a analizar cambios genéticos en los tumores de los pacientes mucho más rápidamente que en la actualidad"
Joe W. Gray, Professor of Laboratory Medicine and Radiation Oncology, University of California, San Francisco, School of Medicine. (JAMA. 1996;275:581-582).
RESUMEN
Las nuevas tecnologías para el tratamiento de la información genética (biochips, sistemas LIMS, bases de datos genómicas, sistemas de minería de datos, técnicas de cuantificación de la expresión génica) están revolucionando la investigación biomédica y se prevé que en los próximos años posibiliten el desarrollo de una nueva práctica médica basada en las particularidades genéticas de los pacientes.
Este nuevo paradigma afecta a la investigación de nuevos fármacos, al diagnóstico clínico, a la salud pública y a la investigación biomédica básica.
La Bioinformática es una disciplina científica que aporta la plataforma tecnológica necesaria para posibilitar estos avances, integrando la información genética con la información clínica, dando respuesta a la necesidad de gestionar elevados volúmenes de información genética en paralelo y proporcionando sistemas deductivos que extraen conocimiento biomédico de utilidad a partir de las bases de datos de investigación y variaciones genéticas individuales.
Desde el Instituto de Salud Carlos III, con la creación de la unidad BIOTIC, especializada en bioinformática aplicada a la salud, se intenta dar respuesta a estos retos, anticipando los cambios que sobrevendrán tras la introducción rutinaria de la información genética en la Salud.
El objetivo de este trabajo es ofrecer una panorámica del estado del arte de estas nuevas tecnologías de tratamiento de la información genética, revisando sus impactos en la investigación biomédica y en la práctica clínica, los aspectos éticos, legales y sociales asociados a su aplicación efectiva en el sistema sanitario para finalizar con una serie de propuestas para tratar de hacer posibles sus beneficios, minimizando los riesgos potenciales.
INTRODUCCIÓN
Genética Molecular
La genética molecular, al igual que los seres vivos, está organizada a modo de escalera en la que hay distintos niveles que están relacionados entre sí y que son capaces de intercambiar información. Se puede considerar el ADN (ácido desoxirribonucleico) como el escalón fundamental que sirve de base para el resto. El ADN está constituido como una doble cadena de nucleótidos que se organizan en una estructura de doble hélice. El ADN es una molécula que porta información agrupada en genes que son secuencias de nucleótidos de longitud variable que sirven para la producción de proteínas. Las moléculas de ADN son extremadamente largas ya que codifican millares de genes (en humanos se estiman alrededor de 80.000-100.000 genes) y en las células aparecen compactadas en forma de cromosomas localizados en los núcleos celulares. El conjunto de todo este material genético presente en los cromosomas recibe el nombre de genoma.
La relación entre el ADN (portador de la información) y las proteínas (resultado de su expresión) se lleva a cabo, mediante una molécula intermedia denominada ARN (ácido ribonucleico). El ADN se transcribe a ARN y éste es traducido en una proteína en otro orgánulo celular denominado ribosoma. Las proteínas se constituyen como polímeros formados por combinaciones de 20 aminoácidos codificados en los genes y son las moléculas que realizan la mayoría de las tareas en las células, tienen papel estructural, actúan como catalizadores de las reacciones químicas, transportan, capturan y almacenan otras moléculas, comunican la célula con el exterior y llevan a cabo la replicación de las moléculas de ADN permitiendo así que la información se conserve de una generación a otra.
Las interacciones moleculares que se dan en el interior celular son complejas y se ven influenciadas de muy diversas maneras por factores internos y externos a las células, desencadenando procesos de expresión génica diferencial. Esta consiste en que, pese a que todas las células de un individuo contienen en su interior la misma información genética, se producen diferencias en la intensidad, momento de la expresión, o en la información expresada. Como resultado de estas variaciones se produce la diferenciación celular y pueden originarse determinadas patologías.
La investigación en genética molecular va a revolucionar la práctica médica y la investigación biológica en el próximo siglo. La localización de los genes humanos permitirá el desarrollo de pruebas diagnósticas fiables para las enfermedades hereditarias y se posibilitará el entendimiento de la influencia de los genes y el medio ambiente en la salud.
Toda esta información genética está produciendo enormes cantidades de mapas complejos y datos de secuencias. Las herramientas informáticas son cruciales para almacenar e interpretar estos datos de un modo eficiente y robusto en los centros de investigación biológica. Estas grandes bases de datos de información biológica son accesibles a través de las redes de comunicaciones, en especial, a través de Internet.
Proyecto Genoma
El progresivo desarrollo de métodos automatizados de preparación de muestras de DNA, su secuenciación y posterior lectura ha permitido afrontar, a lo largo de la ultima década, diversos proyectos de secuenciación a gran escala.
El Proyecto Genoma Humano es el mas ambicioso de todos ellos, constituyéndose como un programa internacional en el que se pretende construir mapas genéticos y físicos detallados del genoma humano, así como determinar su secuencia completa y localizar la posición de los genes que lo forman. Las previsiones parecen apuntar al año 2005, o incluso antes, en cuanto a la fecha de finalización de dicho proyecto.
Algunos datos para entender la magnitud del proyecto:
A medida que el proyecto se acerca a su finalización, el nuevo desafío estriba en conocer la función de todos los genes y su influencia en el desarrollo de las enfermedades.
Bioinformática
La Bioinformática, se encuentra en la intersección entre las Ciencias de la Vida y de la Información, proporcionando las herramientas y recursos necesarios para favorecer la Investigación Biomédica. Este campo interdisciplinar comprende la investigación y desarrollo de sistemas útiles para llegar a entender el flujo de información desde los genes a las estructuras moleculares, a su función bioquímica, a su conducta biológica y, finalmente, a su influencia en las enfermedades y en la salud.
Los estímulos principales para el desarrollo de esta disciplina han sido –el enorme volumen de datos sobre secuencias generados por los distintos proyectos genoma (humano y otros organismos), -los nuevos enfoques experimentales basados en biochips que permiten obtener datos genéticos a gran velocidad, bien de genomas individuales (polimorfismos, mutaciones), bien de enfoques celulares (expresión génica) y el desarrollo de Internet y el WWW, que permiten el acceso universal a las bases de datos de información biológica.
La industria farmaceútica ha sido la primera en darse cuenta de la conveniencia de disponer de personal especializado en bioinformática. La existencia de congresos específicos (ISMB, PSB, RECOMB), sociedades científicas (ISCB), unidades en empresas, centros académicos y del gobierno, empresas dedicadas a prestar servicios en bioinformática, programas de formación en universidades (Oxford, George Mason, Manchester, Pennsylvania, Rutgers, Stanford, Washington-San Luis, CMU, Pittsburgh, Australian National Univ., Pune-India, Dublín, Bergen-Noruega, Johns Hopkins), revistas (Bioinformatics, "In Silico Biology", Trends Guide to Bioinformatics, Journal of Computational Biology), líneas propias de investigación y áreas particulares en convocatorias de proyectos muestran el grado de desarrollo alcanzado por esta disciplina (Gershon, 97, University Bioinformatics Programs).
La Bioinformática en el ISCIII - BIOTIC
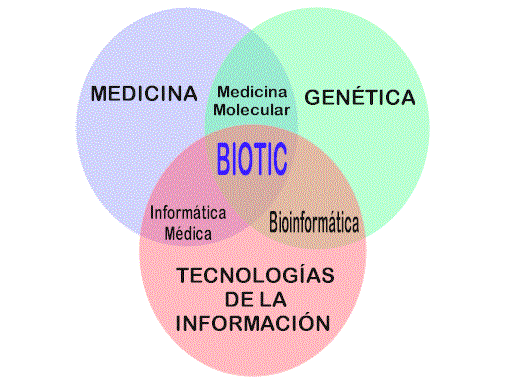
Figura 1. Posicionamiento de la Unidad BIOTIC
El Instituto de Salud Carlos III crea en 1998 la unidad BIOTIC (Aplicaciones en BIOlogía Molecular de las Tecnologías de la Información y las Comunicaciones), con la misión de facilitar el conocimiento de las nuevas tecnologías para el tratamiento de la información genética y su aplicación en la investigación biomédica y la práctica clínica y con la intención de constituirse en centro de referencia en Bioinformática aplicada a la Salud.
Entre sus objetivos se encuentran:
Entre las principales acciones llevadas a cabo por la unidad se pueden destacar la colaboración en biochips con el Centro Nacional de Investigaciones Oncológicas, la impartición de diversos cursos y seminarios de bioinformática a los investigadores del Instituto, así como ponencias en Congresos y conferencias invitadas. Se están suministrando servicios de información sobre genómica en la red interna e internet y existen contactos con la industria farmacéutica para trabajar en la aplicación de las nuevas tecnologías bioinformáticas en el descubrimiento de nuevos fármacos.
NUEVOS ENFOQUES TECNOLÓGICOS PARA EL TRATAMIENTO DE LA INFORMACIÓN GENÉTICA
Biochips
A finales de los años 80, la tecnología que desembocaría en la plataforma GeneChip fue desarrollada por cuatro científicos, en Affymax: Stephen Fodor, Michael Pirrung, Leighton Read y Lubert Stryer. El proyecto original estaba destinado a la construcción de péptidos sobre chips, pero desembocó en la capacidad para construir secuencias de DNA sobre chips. La aplicación práctica de esta idea se llevó a cabo por la empresa Affymetrix, que comenzó a actuar como una compañía independiente en el año 1993.
Los biochips, por tanto, surgieron de la combinación de las técnicas microelectrónicas y el empleo de materiales biológicos. Se basan en la ultraminiaturización y paralelismo implícito y se concretan en chips de material biológico de alta densidad de integración válidos para realizar distintos tipos de estudios repetitivos con muestras biológicas simples.
Si en los microchips empleados en los ordenadores se consigue una alta densidad de integración de circuitos electrónicos en una oblea de silicio, en los biochips se logra una alta densidad de integración de material genético en una oblea de silicio, cristal o plástico.
Los biochips están divididos en unas pequeñas casillas que actúan cada una a modo de un tubo de ensayo en el que se produce una reacción. El número de estas casillas es muy elevado, llegando incluso a los centenares de miles.
Cada casilla del chip posee una cadena de un oligonucleótido, que puede corresponder a una sección del gen de estudio (cuando se conoce su secuencia) o a mutaciones del mismo. Debido a la extrema miniaturización del sistema se pueden analizar en un único chip todas las posibilidades de mutación de un gen simultáneamente. Solo aquellos fragmentos de DNA que hibriden permanecerán unidos tras los lavados y dado que se conocen las secuencias y posiciones de los oligonucleótidos empleados, tras los lavados se produce el revelado que consiste en introducir el chip en un escáner óptico que va a ser capaz de localizar, mediante un proceso similar a la microscopía confocal, las cadenas marcadas con el fluorocromo. Un ordenador analiza la información procedente del escáner y ofrece el resultado. (Wallace, 97).
Otro tipo de diseño permite la cuantificación de la expresión de múltiples genes simultáneamente.
La potencia de estos sistemas trae consigo la obtención, en tiempos muy breves, de grandes volúmenes de información, (secuencias, mutaciones, datos de expresión génica, determinaciones analíticas de interés clínico, screening con fármacos) que necesitan ser gestionados con técnicas bioinformáticas para extraer conocimiento de utilidad en la investigación biomédica.
Parece que el futuro pasa por la integración de estas nuevas técnicas en el entorno clínico haciendo posible el concepto de análisis y diagnóstico en el "point-of-care". La revista Science destaca esta tecnología como uno de los 10 avances científicos más significativos del año 1998 (Bloom, 98).
La nomenclatura empleada para referirse a estas nuevas tecnologías es diversa y comienza por el término más general que es el de "Biochip" y hace referencia al empleo de materiales biológicos sobre un chip. Otros términos más específicos son: "DNA chip", "RNA chip" (según el material empleado) y "Oligonucleotide chip" o "DNA microarray", que hacen referencia al material y a la forma en la que se construye el chip. Existen también unos términos comerciales con los que referirse a los biochips que varían dependiendo de la tecnología empleada.
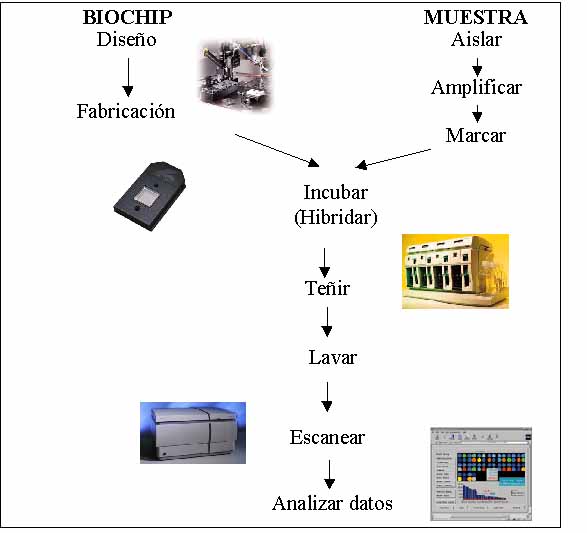
Figura 2. Procedimiento de trabajo con DNA chips (Fuente: Affymetrix – BIOTIC)
Genómica funcional
Si la genómica estructural es la rama de la genómica orientada a la caracterización y localización de las secuencias que conforman el ADN de los genes, la genómica funcional consiste en la recolección sistemática de información sobre la función de los genes, mediante la aplicación de aproximaciones experimentales globales que evalúen la función de los genes haciendo uso de la información y elementos de la genómica estructural. Se caracteriza por la combinación de metodologías experimentales a gran escala con estudios computacionales de los resultados. (Cowley, 98)
Con la genómica funcional el objetivo es llenar el hueco existente entre el conocimiento de las secuencias de un gen y su función, para de esta manera desvelar el comportamiento de los sistemas biológicos. Se trata de expandir el alcance de la investigación biológica desde el estudio de genes individuales al estudio de todos los genes de una célula al mismo tiempo en un momento determinado.
Proteómica
El Proteoma se puede definir como el conjunto de las PROTEinas expresadas por un genOMA. La PROTEOMICA es el estudio de proteomas, así como la GENOMICA consiste en el estudio de genomas. Configura una disciplina fundamental de la era post-genómica que trata de descubrir la constelación de proteínas que otorgan a las células su estructura y función.
Distintas tecnologías permiten obtener y comparar "instantáneas" de las proteínas que se están expresando en un momento determinado en una célula (robótica, electroforesis 2D, espectrometría de masas, chips, bioinformática). (Persidis, 98)
La Nueva Generación de Bioinformática
Se introduce el concepto de Bioinformática de Segunda Generación caracterizada por:
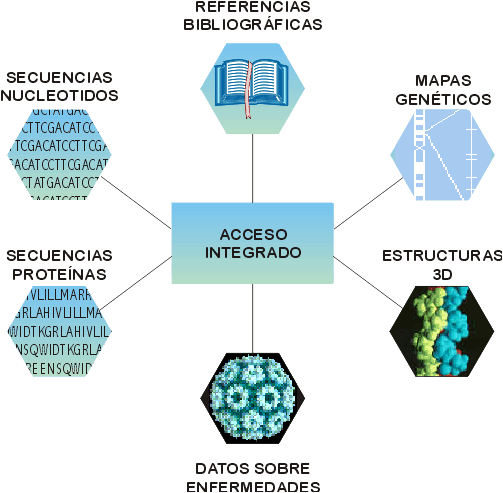
Figura 3. Acceso integrado a información biomédica
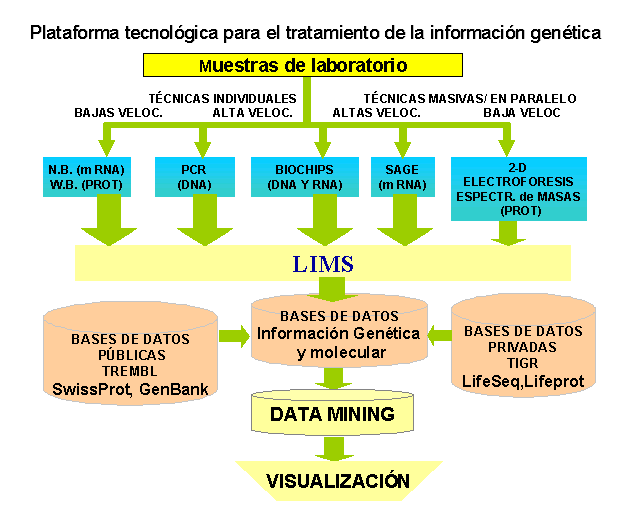
Figura 4. Nuevas tecnologías para el tratamiento de la información genética
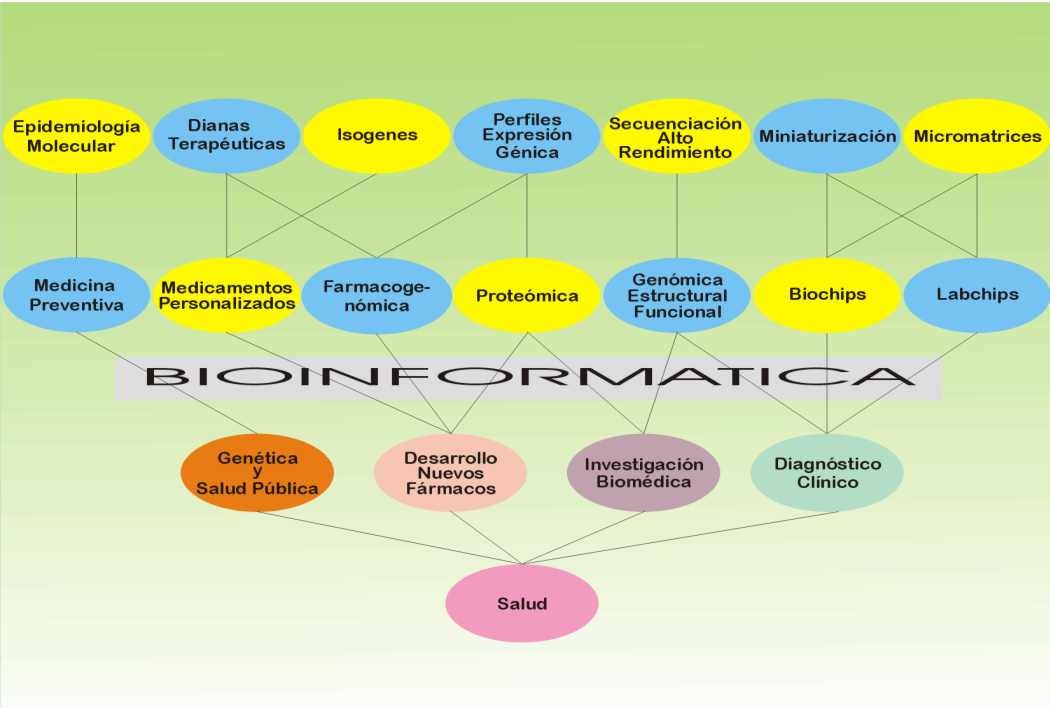
Figura 5. Areas de interés para la nueva bioinformática aplicada a la Salud
IMPACTOS EN LA INVESTIGACIÓN BIOMÉDICA
La aplicación de las nuevas tecnologías de la información genética en el entorno de investigación biomédica ha dado lugar a la aparición de un nuevo vocablo: "Biología in silico": obtención de conocimiento mediante consideraciones teóricas, simulaciones y experimentos llevados a cabo sobre la tecnología basada en silicio de un ordenador. (Wingender, 98). En la figura número 6 se aprecia como las nuevas técnicas permiten que todo el flujo de trabajo de la experimentación biológica sea llevado a cabo de un modo asistido por ordenador.
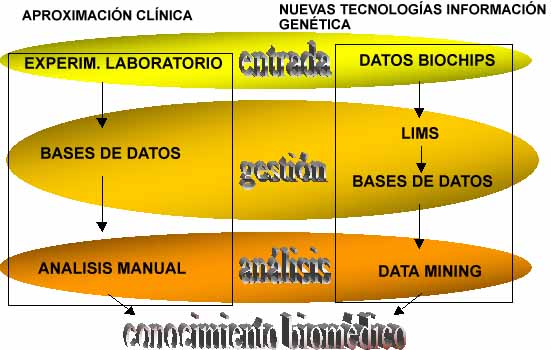
Figura 6. El tratamiento de la información genética. Enfoques clásico y basado en nuevas tecnologías
Desarrollo de fármacos personalizados. Farmacogenómica
¿Porque diferentes pacientes con los mismos síntomas responden de forma diferente a los mismos medicamentos? Aunque el concepto de variaciones individuales a escala molecular no es nuevo, (durante 40 años la farmacología se ha aproximado a este problema desde un punto de vista toxicológico), las plataformas tecnológicas que se proponen para implementar con éxito este concepto se han desarrollado en los últimos 4 años.
Las nuevas tecnologías que ayudan a comprender el papel de los genes en las enfermedades están revolucionando los procesos de descubrimiento y desarrollo de nuevos medicamentos, ofreciendo considerables oportunidades a la industria para reducir tiempos, costes y riesgos. Estamos a punto de asistir al nacimiento de los "medicamentos personalizados" o particularizados para distintos estratos de pacientes clasificados según sus características genéticas. El descubrimiento de las variantes genéticas de los individuos que influyen en el efecto de los fármacos permitirá el desarrollo de nuevos procedimientos diagnósticos y productos terapéuticos que se prescribirán selectivamente a los pacientes, con garantía de seguridad y efectividad.
El empleo de los biochips en screening y toxicología de fármacos permite analizar los cambios de expresión génica que se dan durante la administración de un fármaco de forma rápida así como la localización de nuevas posibles dianas terapéuticas y los efectos toxicológicos asociados (Housman, 98).
El modo en el que se emplea la información genética en el descubrimiento de nuevos fármacos consiste en comparar los genes que se expresan en tejido enfermo y compararlos con los que se expresan en tejido sano, desarrollando medicamentos que bloqueen la acción de los genes que ese encuentran sólo activos en el tejido enfermo.
Polimorfismos y variabilidad de los genomas individuales
Los sistemas de secuenciación rápida permiten el estudio de todos los posibles polimorfismos y la detección de mutaciones en genes complejos. Las secuencias de un mismo gen pueden variar de unos individuos a otros provocando una mutación, que puede producir un cambio en la función de ese gen o ser silenciosa si no produce un cambio funcional. También hay variaciones que aparecen como marcadores del genoma de una determinada estirpe. Los SNPs (single nucleotide polymorphisms) están causados por la mutación de un único residuo de ADN, pudiendo conducir a una pérdida de la función del gen, a una patología o a adquirir susceptibilidad frente a una enfermedad.
El significado de la variación genética humana se analiza observando las mutaciones de secuencias de genes normales y correlacionándolas con enfermedades específicas (Wallace, 97).
Epidemiología Genética y salud pública
La utilización de las nuevas tecnologías de la información genética permitirá la realización de "screening" genéticos coste-efectivos. El reto consiste en trasladar los conocimientos de la genética al campo de la Salud Pública. Para ello hay que asegurar el uso correcto de la información genética y de las nuevas tecnologías que la gestionan en la elaboración de programas de salud. Se habla de sistemas de estudio epidemiológico-genéticos, (genética asociativa, estudios poblacionales genotipo-fenotipo) y se observan esfuerzos encaminados a la diseminación de información genética, la formación de los profesionales de la Salud y el desarrollo de políticas que incorporen el conocimiento genético que se está obteniendo del Proyecto Genoma Humano en la práctica epidemiológica.
La aplicación de las Tecnologías de la Información en esta área permite la realización de esfuerzos colaborativos, como el de la iniciativa HuGeNet (Human Genome Net), de la Oficina sobre Genética y Prevención de Enfermedades del CDC (Khoury, 98). para desarrollar y distribuir información epidemiológica sobre el Genoma Humano, incluyendo:
Expresión génica diferencial
Las tecnologías basadas en biochips permiten estudiar simultáneamente la expresión de miles de genes en un único experimento. Esto posibilita la cuantificación simultánea de la expresión de un número elevado de genes. También permite una aproximación cualitativa comprobando cuál es el patrón de expresión. Se puede estudiar la función de los genes al facilitar la identificación de qué genes están activados de forma diferencial cuando se comparan tejido sano y enfermo (DeRisi, 96), células en distintas etapas de desarrollo, en distintas condiciones metabólicas o ambientales. Estos enfoques posibilitan el desarrollo de la "Genómica Funcional"
Toda la información generada por estos trabajos se almacena en bases de datos especialmente diseñadas para permitir la aplicación de técnicas de minería de datos y visualización para extraer conocimiento biomédico de utilidad.
IMPACTOS EN LA PRÁCTICA CLÍNICA
Por último, hay que anticipar cuales van a ser los principales impactos de la introducción rutinaria de la genética en la práctica médica tal y como hoy la entendemos. Para ello, se describirán los principales conceptos implicados.
Medicina Molecular
Se puede definir como el esfuerzo en explicar la vida y la enfermedad en términos de la presencia y regulación de entidades moleculares.
La práctica médica se verá radicalmente alterada cuando se combine la información genética con las nuevas tecnologías clínicas basadas en el diagnóstico de ADN. Se vislumbra el nacimiento de la "Medicina Molecular", que se caracterizará, no por el tratamiento de los síntomas de las enfermedades, sino por la búsqueda de las causas últimas de estas. Se desarrollarán nuevas técnicas diagnósticas, más rápidas y fiables que facilitarán la prevención de las enfermedades. Se desarrollarán nuevos fármacos que actuarán a escala molecular y personalizada e incluso se practicará la terapia génica, reparando errores genéticos.
Los investigadores ya han identificado genes individuales asociados a varias enfermedades, como la fibrosis cística, la distrofia muscular, la neurofibromatosis y el retinoblastoma. A medida que progrese la investigación genética, se descubrirán los mecanismos que causan enfermedades afectadas por varios genes o por factores ambientales. La predisposición genética está implicada en el desarrollo de enfermedades cardíacas, diabetes y varios tipos de cáncer. La identificación de estos genes y sus proteínas darán lugar a terapias más eficientes y medidas de prevención.
La Medicina Molecular va a permitir redefinir las enfermedades, en lugar de por fenotipo (síntomas), por genotipo y mecanismo causante (distintos tipos de asma), descubrir un origen similar en varias enfermedades (el gen apoE interviene en el origen de enfermedades cardiovasculares y Alzheimer), diseño de nuevos fármacos personalizados y mejorar el seguimiento de terapia, permitiendo la valoración de rasgos genéticos que puedan tener incidencia en la respuesta a la misma, que inviten a variarla o a suprimirla en determinados casos (Hacia, 96).
Medicina preventiva
El conocimiento de los rasgos genéticos de las poblaciones permitiría conocer la predisposición a sufrir algunas enfermedades, antes de que aparezcan síntomas, permitiendo así la realización de una mejor y auténtica medicina preventiva. (Cho, 98). En este entorno la prevención se entiende como la realización de acciones médicas, sobre el medio ambiente o sobre los hábitos de vida, encaminadas a reducir el riesgo de padecer enfermedades por parte de individuos susceptibles por razones genéticas.
Análisis Genético
Se define como el análisis de material genético (ADN, ARN, proteínas, cromosomas), o de ciertos productos metabólicos orientado a la detección de rasgos genéticos y mutaciones heredables, fenotipos o cariotipos. A finales de 1997 ya se disponía de unos 450 análisis para enfermedades genéticas (Sikorski, 97). De ellos, unos 200 se aplican en el estudio genético prenatal.
Los análisis genéticos se emplean para encontrar la posible predisposición de una persona hacia una enfermedad así como para confirmar la sospecha de una mutación en un individuo o en una familia. Sin embargo la creciente atención que esta área despierta se centra en los análisis predictivos, que identifican a las personas con alto riesgo de contraer una enfermedad, antes de que los síntomas asociados aparezcan. Ya existen investigaciones para ensayos en unas 20 enfermedades y a medida que se descubren más genes, se puede esperar la aparición de otros análisis. Hay que asegurar la seguridad y efectividad de estas técnicas de análisis genético, no sólo en sus aspectos de utilidad, sino de aplicación en el laboratorio, y de uso apropiado por parte de pacientes y profesionales.
Diagnóstico en Microbiología
El diagnóstico basado en la detección de microorganismos permite la identificación rápida empleando unos marcadores genéticos de los patógenos, así como de los posibles mecanismos asociados a la patogenicidad. Su finalidad es la de permitir la comprensión de la biología de los microorganismos, estudiar los mecanismos de resistencia frente a antibióticos, identificación de las cepas, identificar nuevas dianas génicas con valor terapéutico y el desarrollo de medidas preventivas frente a las enfermedades infecciosas (Gingeras, 98; Harby, 96).
INFORMÁTICA MÉDICA Y BIOINFORMÁTICA
La Informática Médica y la Bioinformática están acercándose, a medida que se genera información del Genoma Humano y se enlaza con el conocimiento médico sobre las enfermedades. (Altman, 96). Esta tendencia se aprecia en el hecho de que Congresos como MIE, AMIA o MEDINFO tienen sesiones específicas sobre bioinformática y los máster de informática médica de varias universidades, como Stanford, incluyen formación en bioinformatica (Shortliffe, 98).
Dado el enorme ritmo de generación de conocimiento médico basado en la genética, se puede pensar que en unos años, el médico tendrá que acudir a Internet para encontrar la información actualizada que necesita para su práctica habitual (Sikorski, 97).
Conseguir unir la información clínica con la información molecular representa uno de los retos más importantes del próximo siglo para la Bioinformática. (Altman, 98). Los datos que maneja la bioinformática van a tener cada vez más presencia en la práctica médica.
Aunque la bioinformática y la informática médica comparten muchas metodologías, tipos de datos y algoritmos, se puede destacar que la bioinformática está más avanzada en:
La informática médica ha alcanzado mayor desarrollo en áreas como:
Existen un buen número de áreas de interés para ambas, donde puede existir sinergia:
Historia clínica
En la base de datos OMIM hay más de 4.000 genes asociados a enfermedades. Ya existen pruebas genéticas para varias (Huntington, fibrosis cística, cáncer). Aunque actualmente los análisis se llevan a cabo en laboratorios centrales y tardan varios días, ya existen tecnologías que permiten poner un laboratorio genético en la consulta del médico general, e incluso en las farmacias.
Esto es así porqué se ha pasado de la secuenciación basada en PCR a los nuevos biochips, que nos permiten aproximarnos al concepto de un "sistema automatizado para genotipado clínico".
El resultado de los biochips está informatizado – esa información se integrará en la Historia clínica aportando las siguientes ventajas:
Todo apunta a que, en unos años, (Altman, 98bis, Naser, 98) la información genética se incluirá de modo rutinario en la historia clínica informatizada.
Impactos en la gestión sanitaria
Las guías de práctica clínica deberán incluir aspectos genéticos, se introducirán nuevos sistemas de codificación, las terapias y los fármacos se prescribirán de modo personalizado, los médicos de atención primaria dispondrán de herramientas para el diagnóstico genético rápido en la propia consulta y los gestores tendrán que asegurar el acceso de los médicos a recursos genéticos on-line, principalmente vía internet.
DISCUSIÓN. ASPECTOS ÉTICOS, LEGALES Y SOCIALES DEL USO DE LA INFORMACIÓN GENÉTICA EN SALUD
Existen muchos beneficios y riesgos potenciales en la aplicación de estas tecnologías en la realidad sanitaria. Entre los impactos de las tecnologías de adquisición y gestión de la información genética, aplicadas en la investigación y práctica biomédica, se pueden citar:
La información generada por el Proyecto Genoma es sólo la punta del iceberg. Vamos a asistir a una explosión de datos a medida que se extiendan las tecnologías de la información genética (biochips, obtención de genomas individuales, proteómica, datos de expresión génica). Se necesitan herramientas que asistan al investigador y al clínico en el acceso eficiente a las fuentes de datos, en su interpretación y a la hora de aplicarlas en el entorno sanitario (Thornton, 98, Boguski, 98).
Las tecnologías basadas en biochips pueden hacer por la genética lo que los microprocesadores hicieron por la informática. La miniaturización alcanzada permitirá que el diagnóstico salga de los laboratorios centrales y llegue hasta la consulta del médico, del mismo modo que los ordenadores pasaron de estar sólo en los centros de cálculo a la ubicuidad actual de los ordenadores personales. (Gibbs, 96).
Para finalizar se apunta desde aquí la necesidad de debates sociales informados sobre estos impactos de la nueva "Medicina individualizada", que forzosamente tendrán que reparar en aspectos relacionados con la exactitud de estas técnicas, con la seguridad y privacidad de la información generada y con la necesidad de incrementar el conocimiento genético de los pacientes para que puedan entender el uso que se hace de sus datos y de los profesionales de la salud para que presten adecuado consejo genético e interpreten correctamente las pruebas y con los esfuerzos legislativos encaminados a impedir la discriminación de origen genético y el mal uso de la información por parte de la sociedad.
BIBLIOGRAFÍA
Altman, R.B., Bioinformatics in support of molecular medicine. En C.G.Chute, Ed., AMIA Annual Symposium, Orlando, 1998. p. 53-61.
Altman, R.B., A curriculum for bioinformatics: The time is ripe. Bioinformatics, 1998. 14, 7, p. 549-550.
Altman, R.B. & Koza, J., A programming course in bioinformatics for computer and information Science students. En L. and Klein Hunter T.E., Ed. Pacific Symposium of Biocomputing´96, Hawaii, 1996. p. 73-84. World Scientific, Singapore.
Bloom, F.E. Breakthroughs 1998. Science, 1998. 282(5397): p. 2193-2161
Boguski, M.S., Bioinformatics-A new era. Trends guide to Bioinformatics, 1998. 1: p.1-3.
Cho, R.J., et al., Parallel analysis of genetic selections using whole genome oligonucleotide arrays. Proc Natl Acad Sci U S A, 1998. 95(7): p. 3752-7.
Cowley, A., W., The Emergence of Physiological Genomics INABIS 98 - 5th Internet World Congress on Biomedical Sciences. 1998, Canadá.
DeRisi, J., et al., Use of a cDNA microarray to analyse gene expression patterns in human cancer. Nat Genet, 1996. 14(4): p. 457-60.
Ermolaeva, O., et al., Data management and analysis for gene expression arrays. Nat Genet,1998. 20(1): p.19-23
Gershon, D., Bioinformatics in a post-genomics age. Nature, 1997. 389: p. 417-418.
Gibbs, W.W., New chip off the old block. Can DNA microprobes do for genetics what microprocessors did for computing? [news]. Sci Am, 1996. 275(3): p. 42, 44.
Gingeras, T.R., et al., Simultaneous genotyping and species identification using hybridization pattern recognition analysis of generic Mycobacterium DNA arrays. Genome Res, 1998. 8(5): p. 435-48.
Hacia, J.G., et al., Detection of heterozygous mutations in BRCA1 using high density oligonucleotide arrays and two-colour fluorescence analysis. Nat Genet, 1996. 14(4): p. 441-7.
Harby, K., Genes on a chip for tissue expression and HIV study [news]. Mol Med Today, 1996. 2(8): p. 317.
Housman, D. et al., Why pharmacogenomics? Why now? Nat. Biotechnol, 1998. 16(6): p. 492.
Khoury, M. J., HuGENet. 1st Annual Conference on Genetics and Public Health. 1998.
http://www.cdc.gov/genetics/publications/abstracts.html
Martín-Sánchez, F., López-Campos, G., Tecnologías basadas en Biochips. Aplicaciones en diagnóstico clínico e investigación biomédica. II Symposium Internacional sobre Diagnóstico Genético en Medicina. 1998. Madrid.
Michaud, G. Y., The FDA Review of "in vitro" diagnostic devices, an overview. 1st Annual Conference on Genetics and Public Health. 1998. http://www.cdc.gov/genetics/publications/abstracts.html
Naser, C., R., High Speed Genetic Testing Technology and the Computerization of the Medical Record. 1998 http://www.math.luc.edu/ethics97/papers/Naser.html
Persidis, A., Proteomics. Nat Biotechnol, 1998. 16(4): p. 393
Sikorski, R.& Peters, R., Genomic Medicine, internet resources for medical genetics. JAMA 1997. 278(15): p.1212-1213
Shortliffe, E.H., Computational biology and the cross-disciplinary challenge: finding a home in academia. 1998. May. Computer Science and Telecommunications Board, Natl. Res. Council. Tenth Aniversary Symposium.
Thornton, J.M., The future of Bioinformatics. Trends guide to bioinformatics, 1998. 1: p.30-31.
University Bioinformatics Programs.
http://biotech.chem.indiana.edu/pages/bioinform/biprograms_us.htm
Wallace, R.W., DNA on a chip: serving up the genome for diagnostics and research. Mol Med Today, 1997. 3(9): p. 384-9.
Wingender, E., In Silico Biology: Just another Journal? In Silico Biology, 1998. 1.
