 |
 |
[Entrada] [Actividades]
[Revista I + S] [Solicitud de
Inscripción]
BIOINFORMÁTICA, GENÓMICA Y SALUD
Importancia de la Bioinformática y la Genómica en el nuevo Plan Nacional de I+D+I
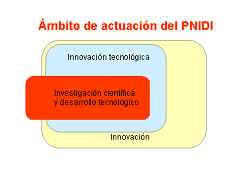
El Plan Nacional de Investigación Científica, Desarrollo e Innovación Tecnológica (PN) para el periodo 2000-2003, se ha concebido bajo una óptica integradora y pretende definir una estrategia global que incluya todas las actuaciones públicas gestionadas por Departamentos ministeriales con competencias en I+D. El PN se estructura en torno a un número limitado de áreas de actividad prioritarias que son elementos clave para su definición y que lo configuran de acuerdo con la estructura global y los objetivos estratégicos del mismo. Estas áreas concuerdan con las que se contemplan en los programas internacionales, especialmente, a escala europea, y además se han tenido en cuenta criterios estratégicos, históricos y culturales de la sociedad española.
La Biomedicina y Biotecnología constituyen dos áreas prioritarias científico tecnológicas. El desarrollo en ambas áreas está estrechamente relacionado. En ambas áreas se pretende potenciar la investigación genómica y postgenómica así como de la bioinformática, herramienta imprescindible para el desarrollo de estas.
Debido al extraordinario avance de la genética molecular y la genómica, la Biomedicina consta como arma estratégica del bienestar social del futuro inmediato. Se pretende potenciar la aplicación de las nuevas tecnologías y de los avances genéticos para el beneficio de la salud. Dentro de las actividades financiables además de los proyectos I+D, existen acciones estratégicas, de infraestructura, centros de competencia y grandes instalaciones científicas. En esta área, la dotación de infraestructura se plasmará en la creación y dotación de unidades de referencia tecnológica y centros de suministro común, como centros de bioinformática, que cubran las necesidades de la investigación biomédica. En cuanto a centros de competencia, se crearán centros de investigación de excelencia en hospitales en los que se acercará la investigación básica a la clínica, así como centros distribuidos en red para el apoyo a la secuenciación, DNA microarrays y DNA chips, bioinformática, en coordinación con la red de centros de investigación genómica y proteómica que se proponen en el área de Biotecnología. En este área la genómica y proteómica constan como acción estratégica o instrumento básico de focalización de las actuaciones del PN, cuyos resultados pretenden ser alcanzados dentro del periodo cubierto por el PN.
Las tecnologías de la información jugarán un papel fundamental en la aplicación de los desarrollos tecnológicos en el campo de la genética a la práctica médica como refleja la presencia de la Bioinformática médica y la Telemedicina dentro de las principales líneas de I+D en Biomedicina.
La aplicación de los conocimientos en genética molecular y las nuevas tecnologías son necesarios para el mantenimiento de la competitividad del sistema sanitario no sólo paliativo sino preventivo. La identificación de las causas moleculares de las enfermedades junto con el desarrollo de la industria biotecnológica en general y de la farmacéutica en particular permitirán el desarrollo de mejores métodos de diagnóstico, la identificación de dianas terapéuticas y desarrollo de fármacos personalizados y una mejor medicina preventiva.
IBM se une al consorcio que está realizando el mapa de la diversidad genética humana
Dentro de la línea de actuación que se ha estado detectando en las grandes empresas de las tecnologías de la información, la multinacional IBM anunció en el pasado mes de Enero su intención de participar en un consorcio internacional creado con la finalidad de establecer un mapa de marcadores genéticos humanos para el año 2001.
El consorcio internacional, que está formado por compañías farmacéuticas y centros académicos, cuenta con un presupuesto de cincuenta millones de dólares, y tiene ahora un nuevo socio que va a aportar tres millones de dólares y que simboliza la importancia que estas grandes compañías de las tecnologías de la información están dando a las aplicaciones existentes en el campo de las ciencias biomédicas.
El SNP Consortium, que es el nombre con el que se ha bautizado a este proyecto está constituido por prestigiosos organismos de investigación como el Wellcome Trust y grandes compañías farmacéuticas, como Bayer AG. , Novartis y las recientemente fusionada Glaxo-SmithKline. Ya contaba también con la presencia de otra gran empresa de las tecnologías de la información como es Motorola. Ahora con la entrada dentro de este proyecto de este nuevo socio tecnológico se espera impulsar más aún su desarrollo y el beneficio de los demás socios participantes. La finalidad de este consorcio es como ya se ha dicho anteriormente la identificación de los 300.000 SNP´s (Single Nucleotides Polymorphisms) que se estiman están presentes en el genoma humano y la elaboración de un mapa en el que se sitúe cada uno de estos polimorfismos, que será públicamente accesible a través de la bases de datos dbSNP (
http://ncbi.nlm.nih.gov/SNP).La entrada de IBM proporcionará al consorcio otro socio capaz de aportar la tecnología necesaria para resolver e impulsar las soluciones y requerimientos bioinformáticos que son necesarios a la hora de plantearse abordajes tan complejos como son los objetivos de este proyecto.

En palabras del vicepresidente para ciencias de la vida de IBM, "las ciencias de la vida significarán para las primeras décadas de este siglo lo que los semiconductores fueron a las últimas décadas del siglo que acabamos de despedir". En esta misma dirección se expresó el director del departamento para el desarrollo de soluciones para las ciencias de la vida al referirse a la necesidad que presenta la genómica de un incremento en al potencia de los ordenadores actuales así como de un mayor desarrollo en los aspectos informáticos.
La actuación de IBM, al entrar en este consorcio, continúa demostrando su interés por el sector biomédico, reforzado por otras iniciativas ya anunciadas tales como la creación de una división para las ciencias de la vida o la construcción en cinco años de un superordenador con capacidad del orden del petaflop (noticia publicada en el anterior número de I+S) y destinado a la elucidación de estructuras proteicas. IBM también está participando en la iniciativa de genómica estructural propuesta por el NIH, ofreciendo fondos por valor de un millón de dólares para la utilización de superordenadores IBM RS/6000 SP y otras tecnologías permitiendo también el acceso de los centros implicados a software y otros recursos en el Deep Computing Institute de esta empresa.
Affymetrix anuncia la efectividad de la adquisición de Genetic MicroSystems (GMS) y la comercialización de dos nuevos GeneChips
Pese a que la empresa estadounidense Affymetrix, una de las empresas líderes en el mercado de los biochips, anunció en el pasado otoño la compra de Genetic Microsystems por un montante de 120 millones de dólares, la operación no se ha hecho efectiva hasta el mes de Febrero. Con esta operación, Affymetrix se consolida en la posición de liderazgo que tenía dentro del mercado de los biochips y representa una referencia obligada por ser considerada como una de las creadoras del concepto actual de los biochips.
Genetic Microsystems era una empresa dedicada a la fabricación y comercialización de "arrayers", que es el nombre que reciben los robots que permiten la fabricación de biochips por parte de los investigadores en los laboratorios y escáneres para la detección y revelado de los ensayos con biochips.
Affymetrix estaba anteriormente centrada en la fabricación industrial y posterior comercialización de los biochips conocidos como GeneChips, así como en el desarrollo del software necesario para desarrollar los experimentos, realizar los análisis y almacenar los resultados. Con la absorción de GMS por parte de Affymetrix, esta última logra alcanzar una posición destacada en el mercado desde la cual será capaz ahora de atender a todos los sectores de demanda de estas tecnologías, al tener acceso ahora a los sistemas fundamentados en el "hágalo usted mismo" que es el concepto en el que subyacen los "arrayers".
A partir de ahora los equipos que anteriormente llevaban el distintivo de GMS lo verán sustituido por el logotipo de Affymetrix y cambiarán sus nombres sustituyendo GMS por Affymetrix.
La empresa Affymetrix también ha anunciado durante este mes de Febrero la salida al mercado de dos nuevos chips, el GeneChip Human Cancer G110 y el GeneChip Yeast Genome S98.

El chip Human Cancer G110, es un chip que contiene 1.700 genes inmovilizados y que han sido seleccionados por un panel de expertos a los que reunió la empresa para determinar cuales son los genes más relevantes en la investigación del cáncer. El chip que ahora se comercializa será empleado por los investigadores para analizar las variaciones que se produzcan en la expresión génica bajo diferentes circunstancias permitiendo profundizar en el conocimiento de la función de estos genes y con posterioridad asociarlos a distintas rutas metabólicas y de enfermedades.
El otro chip del que se anunció su salida al mercado, el GeneChip Yeast Genome S98, es un chip desarrollado para permitir el análisis completo de la expresión génica en levaduras y que supone una actualización sobre el conjunto Ye6100 que también permitía analizar simultáneamente la expresión del genoma completo de este microorganismo. Las principales diferencias entre los dos productos se centran en que el Yeast Genome S98 contiene sondas que permiten la identificación de genes recientemente descubiertos, y la más importante de ellas es que ha sido fabricando empleando los últimos avances tecnológicos, permitiendo que en la actualidad se pueda contener todo el genoma de la levadura en un único chip en lugar de los cuatro que empleaba su predecesor.
Aplicación de los estudios de expresión génica basados en biochips para la clasificación de linfomas
En el número de la revista Nature (403:503-11) publicado el 3 de febrero último aparece un artículo firmado entre otros por P.O. Brown, que es considerado como uno de los gurús en el terreno de los biochips, en el cual se explica la aplicación de este tipo de dispositivos en diagnóstico molecular del cáncer.
Los ensayos se llevaron a cabo en el linfoma diseminado de gran extensión (DLBCL), que es el linfoma de tipo no-Hodgkin más común. Para la realización de este estudio ha sido necesario el diseño y fabricación de biochips específicos y que se han conocido como "Lymphochips". En el diseño de este Chip se emplearon 17.856 clones de cDNA de genes con una expresión preferencial en tejido linfoide de los que se conoce o se cree que pueden jugar un papel importante en los procesos inmunológicos u oncogénicos.
El estudio consistió en el análisis de los patrones de expresión de tres alteraciones linfoides malignas comparados con los patrones de expresión de diferentes subpoblaciones linfoides normales purificados en diferentes estados de activación, y diversas líneas celulares de linfomas y leucemias. Tras un complejo proceso de análisis de los datos, los resultados condujeron al descubrimiento de dos subtipos de DLBCL en función de diferencias detectadas en sus patrones de expresión génica. En uno de estos subtipo las células presentaban un patrón de expresión característico de las células B de los centros germinales y en el otro caso se asemejaba más al patrón de las células B activadas. El descubrimiento de estos dos subgrupos impulsó la investigación de la posible relevancia clínica y la posible definición de dos subgrupos de pacientes en función del subtipo DLBCL. Para ello emplearon registros de pacientes a los que se les había diagnosticado esta patología, encontrando que una de las formas, la que presentaba un patrón similar al de las células B activadas, presentaba un peor pronóstico a cinco años que la forma con un patrón similar al de los centros germinales.
Los autores señalan que las dos formas de linfoma se ven acompañadas por grandes diferencias en el comportamiento clínico por lo que deberían ser consideradas como diferentes enfermedades.
La publicación de este ensayo viene a resaltar las posibles aplicaciones de los biochips en la práctica clínica, lo que unido a los bajos costes que pueden tener estos dispositivos puede conducir a su extensión en un futuro sustituyendo a muchos de los ensayos actuales.
Establecimiento de unas guías de procedimiento en Genómica Estructural
La posibilidad del establecimiento de un proyecto internacional en genómica estructural pudiera no estar tan lejos a la luz de las reuniones que se están llevando a cabo entre científicos de Estados Unidos, Reino Unido, Alemania, Francia, Israel y Japón, y que están dirigidas hacia el establecimiento de unos procedimientos en el trabajo de la Genómica Estructural. Este proyecto podría ser tan ambicioso, de llevarse a cabo, como lo fue en su día el Proyecto Genoma Humano.
En el próximo mes de Abril se celebrará una reunión en el Sanger Centre (Reino Unido), en la que se tratarán las reglas de colaboración y las políticas de liberación de la información y los derechos intelectuales de los hallazgos que se realicen durante las investigaciones.
La finalidad de la genómica estructural es catalogar las propiedades del plegamiento de todas las proteínas naturales. Las similitudes presentes a nivel de la secuencia génica pueden ser similitudes en la estructura de la proteína codificada, con lo cual se puede hacer una clasificación de las formas proteicas en familias. Esta clasificación reduciría el tiempo y coste necesario para la realización de estudios para conocer la estructura de las proteínas.
A diferencia de los estudios de secuenciación, el análisis y unificación de un conjunto de proteínas presenta mayores dificultades técnicas que el mismo trabajo con ácidos nucleicos. Las tareas de secuenciación de los genomas están altamente automatizadas, mientras que el trabajo con proteínas resulta mucho más complejo, presentando una mayor cantidad de cuellos de botella. Por lo tanto se antoja el fraccionamiento de este trabajo entre los grupos como el mecanismo que posibilitaría la realización de este proyecto.
La determinación de los puntos de inicio y finalización de un proyecto semejante resultan complejas, dadas algunas dificultades técnicas como por ejemplo la dificultad de cristalización de las proteínas presentes en las membranas celulares lo que imposibilita su análisis cristalográfico y por tanto la elucidación última de la estructura.
En la próxima reunión se tratarán también las necesidades en los estándares que deben cumplir los datos antes de ser aceptados por una base de datos central; los tiempos; cuales son las principales proteínas de cada grupo y el reparto de tareas.
Desde Junio del pasado año en Estados Unidos y a propuesta del Instituto Nacional da Salud (NIH) se ha puesto en marcha una iniciativa similar, que cuenta con un programa piloto de una duración estimada de 5 años y en la que se espera conseguir la elucidación de un gran número de estructuras de proteínas, del mayor número de familias posibles. Otro de los grandes objetivos de este programa es el desarrollo de nuevos métodos y tecnologías que mejoren la eficiencia de las técnicas de determinación de estructuras mediante aproximaciones masivas y la automatización de los procesos.
Uno de los mayores problemas que se pueden crear como consecuencia del impulso que se está dando con estos programas a la genómica estructural es la duplicidad de los esfuerzos tanto nacional como internacionalmente lo que conllevaría un mal aprovechamiento de los recursos, aún así en palabras de S. Burley, profesor de la Rockefeller University e investigador del hospital Howard Hughes "la existencia de cierto solapamiento resulta positiva al permitir el control de la calidad, pero en exceso resulta perjudicial y un derroche".
