

|
DETECCIÓN DE HIPERMETILACIÓN DE GENES SUPRESORES TUMORALES EN ASTROCITOMAS Aiala Lorente*, Paula Lazcoz**, Jose Maria Rivera***, Jesus Garibi***, Javier S. Castresana* |
|
|
INTRODUCCIÓN Los astrocitomas son el tipo de tumor intracraneal más frecuente, característico por invadir las regiones cerebrales próximas. Se clasifican en los siguientes tipos, de acuerdo con la OMS: Grado I, Astrocitoma pilocítico; Grado II, Astrocitoma difuso de bajo grado; Grado III, Astrocitoma anaplásico; Grado IV, Glioblastoma multiforme (GBM), que puede ser primario, formado de novo o secundario formados a partir de tumores de grado II y III por progresión tumoral. Se han estudiado los siguientes genes supresores tumorales: PTEN (10q23.3); MGMT (10q26.1); dos efectores de Ras: RASSF1A (3p21.3) y NORE1A (1q32.1); BLU (3p21.3); y CDKN2A (9p21) que codifica dos proteínas alternativas: p14ARF y p16INK4A. La hipermetilación de las islas CpG de promotores puede silenciar estos genes, de manera que disminuya su expresión, contribuyendo a la génesis de diversos tipos de tumores. MATERIAL Y MÉTODOS El trabajo se realizó sobre 53 muestras humanas procedentes del Hospital de Cruces de Barakaldo (España) de astrocitomas de diversos grados y 10 líneas celulares de astrocitoma. La detección de hipermetilación de promotores: se realizó mediante MSP (PCR específica de metilación) para tumores y líneas. El estudio de expresión: se realizó por RT-PCR en RNA obtenido de las líneas. RESULTADOS Detección de hipermetilación de promotores (porcentaje de muestras hipermetiladas halladas): Muestras tumorales: RASSF1A: 92% (49 de 53), BLU: 51% (27 de 53), NORE1A: 4% (2 de 52), PTEN: 0% (0 de 51), MGMT: 47% (25 de 53), p14: 0% (0 de 53), p16: 25% (13 de 53). Líneas celulares: RASSF1A: 100% (10 de 10), BLU: 33% (3 de 9), NORE1A: 40% (4 de 10), PTEN: 20% (2 de 10), MGMT: 80% (8 de 10), p14: 0% (0 de 5), p16: 29% (2 de 7). CONCLUSIONES El gen RASSF1A presentó un alto grado de hipermetilación, tanto en los tumores (92%) como en las líneas (100%), que se correspondió con una expresión baja o nula de dicho gen en la mayoría de las líneas celulares, indicando que podría ser un evento frecuente en la génesis de los astrocitomas. La hipermetilación de BLU, NORE1A, p16 y MGMT parece contribuir también, aunque en menor medida, mientras que la de p14 y PTEN parece no estar implicada, ya que no se observó en ningún caso.
|
||
|
|
Los astrocitomas son el tipo de tumor intracraneal más frecuente,
característico por invadir las regiones cerebrales próximas. Se
clasifican en los siguientes tipos, de acuerdo con la OMS: Grado I,
Astrocitoma pilocítico; Grado II, Astrocitoma difuso de bajo grado;
Grado III, Astrocitoma anaplásico; Grado IV, Glioblastoma multiforme
(GBM), que puede ser primario, formado de novo o secundario formados a
partir de tumores de grado II y III por progresión tumoral. Se han estudiado los siguientes genes supresores tumorales: PTEN (10q23.3); MGMT (10q26.1); dos efectores de Ras: RASSF1A (3p21.3) y NORE1A (1q32.1); BLU (3p21.3); y CDKN2A (9p21) que codifica dos proteínas alternativas: p14ARF y p16INK4A. La hipermetilación de las islas CpG de promotores puede silenciar estos genes, de manera que disminuya su expresión, contribuyendo a la génesis de diversos tipos de tumores.
|
|
|
|
El trabajo se realizó sobre 53 muestras humanas procedentes del
Hospital de Cruces de Barakaldo (España) de astrocitomas de diversos
grados y 10 líneas celulares de astrocitoma. La detección de hipermetilación de promotores: se realizó mediante MSP (PCR específica de metilación) para tumores y líneas. El estudio de expresión: se realizó por RT-PCR en RNA obtenido de las líneas.
|
|
|
|
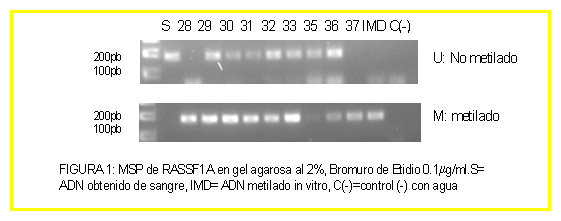
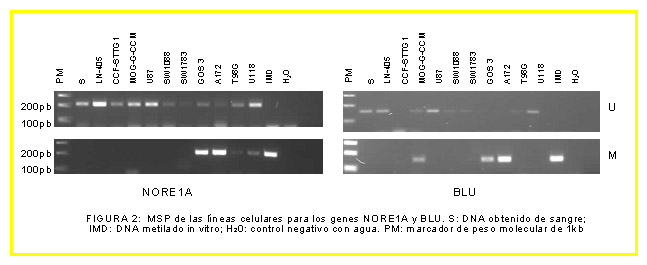
Detección de hipermetilación de promotores (porcentaje de muestras hipermetiladas halladas): Muestras tumorales: RASSF1A: 92% (49 de 53), BLU: 51% (27 de 53), NORE1A: 4% (2 de 52), PTEN: 0% (0 de 51), MGMT: 47% (25 de 53), p14: 0% (0 de 53), p16: 25% (13 de 53). Líneas celulares: RASSF1A: 100% (10 de 10), BLU: 33% (3 de 9), NORE1A: 40% (4 de 10), PTEN: 20% (2 de 10), MGMT: 80% (8 de 10), p14: 0% (0 de 5), p16: 29% (2 de 7).
|
|
|
|
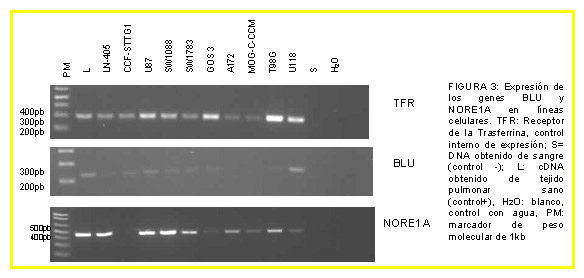
El gen RASSF1A presentó un alto grado de hipermetilación, tanto en los
tumores (92%) como en las líneas (100%), que se correspondió con
una expresión baja o nula de dicho gen en la mayoría de las líneas
celulares, indicando que podría ser un evento frecuente en la génesis
de los astrocitomas. La hipermetilación de BLU, NORE1A, p16 y MGMT parece contribuir también, aunque en menor medida, mientras que la de p14 y PTEN parece no estar implicada, ya que no se observó en ningún caso.
|
|
|
|
1. Kleihues, P.a.C.W., Pathology and Genetics of Tumors of the Nervous System, ed W.H.O. C.o. Tumours. 2000, Lyon. 2. Van Meyel DJ, Ramsay DA, Casson AG, Keeney M, Chambers AF, Cairncross JG. p53 mutation, expression, and DNA ploidy in evolving gliomas: evidence for two pathways of progression. J Natl Cancer Inst 1994; 86: 1011-1017. 3. Robertson KD. DNA methylation, methyltransferases, and cancer. Oncogene, 2001; 20(24): 3139-55. 4. Somerville RP, Shoshan Y, Eng C, Barnett G, Millar D, Cowell JK. Molecular análisis of two putative tumour supresor genes, PTEN and DMBt, which have been implicated in glioblastoma multiforme disease progression. Oncogene 1998; 17:1755-7. 5. Maehma T, Dixon JE. The tumor suppressor gene, PTEN/MMAC1, desphosphorilates the lipid second messenger, phosphatidylinositol 3,4,5-trisphosphate. J Biol Chem 1998; 273:13375-8. 6. Myers MP, PassI, Batty IH et al. The lipid phosphatase activity of PTEN is critical for its tumor suppressor function. Proc Natl Acad Sci USA 1998;95:13513-8 7. Wen S, Stolarov J, Myers MP et al. PTEN controls tumor-induced angiogenesis. Proc Natl Acad Sci USA 2001; 98:4622-7. 8. EstellerM, Hamilton SR, Burger PC, Baylin SB, Herman JG. Inactivation of the DNA repair gene o6-methylguanine-DNA methyltransferase by promoter hypermethylation is a common event in primary human neoplasia. Cancer REs 1999; 59:793-7. 9. Dammann R, Li C, Yoon JH, chin PL, Bates S, Pfeifer GP. Epigenetic inactivation of a RAS association domain family protein from the lung tumour suppressor locus 3p21.3. Nat Genet 2000; 25:315-9. 10. Song MS, Song SJ, Ayad NG et al. The tumour suppressor RASSF1A regulates mitosis by inhibiting the AP-Cdc20 complex. Nat Cell Biol 2004; 6:129-37. 11. Hesson L, Bièche I, Krex D, Criniere E, Hoang-Xuan K, Maher ER, Latif F. Frequent epigenetic inactivation of RASSF1A and BLU genes located within the critical 3p21.3 region in gliomas. Oncogene 2004; 23, 2408-19 12. Horiguchi K, Tomizawa Y, Tosaka M, Ishiuchi S, Kurihara H, Mori M and Saito N. Epigenetic inactivation of RASSF1A candidate tumor suppressor gene at 3p21.3 in brain tumors. Oncogene 2003; 22, 7862-7865 13. Harada K, Toyooka S, Maitra A, Maruyama R, Toyooka KO, Timmons CF, Tomlinson GE, Mastrangelo D, Hay RJ, Minna JD, Gazdar AF. Aberrant promoter methylation and silencing of the RASSF1A gene in pediatric tumors and cell lines. Oncogene 2002; 21: 4345-49. 14 Lusher ME, Lindsey JC, Latif F, Pearson ADJ, Ellison DW, Clifford SC. Biallelic epigenetic inactivation of the RASSF1A tumor suppressor gene in medulloblastoma development. Cancer Research 2002; 62: 5906-11. 15. Burbee DG, Forgacs E, Zöchbauer-Müller S, Shivakumar L, Fong K, Gao B, Randle D, Kondo M, Virmani A, Bader S, Sekido Y, Latif F, Milchgrub S, Toyooka S, Gazdar AF, Lerman MI, Zabarovsky E, White M, Minna JD. Epigenetic inactivation of RASSF1A in lung and breast cancers and malignant phenotype suppression. Journal of the National Cancer Institute 2001; 93:691-99. 16. Ortiz-Vega S, Khokhlatchev A, Nedwidek M et al. The putative tumor suppressor RASSF1A homodimerizes and heterodimerezes with the Ras-GTP binding protein Nore1A. Oncogene 2002; 21:1381-90 17 Hesson L, Dallol A, Minna JD, Maher ER, Latif F. NORE1A, a hologue of RASSF1A tumour suppressor gene is inactivated in human cancers. Oncogene 2003; 22:947-54 18. Morris MR, Hesson LB, Wagner KJ, et al. Multigene methylation analysis of Wilms tumour suppressor gene is inactivated in human cancers. Oncogene 2003;22:6794-801. 19. Agathanggelou A, Dallol A, Zöchbauer-Müller S, Morrissey C, Honorio S, Hesson L, Martinsson T, Fong KM, Kuo MJ, Yuen PW, Maher ER, Minna JD and Latif F Epigenetic inactivation of the candidate 3p21.3 suppressor gene BLU in human cancers. Oncogene 2003; 22, 1580-1588 20. Fan X, Muñoz J, Sanko SG, Castresana JS Pten, DMBT1 and p16 alterations in diffusely infiltrating astrocytomas. Int J Oncol 2002; 21: 667-74 21 Moulton T, Samara G, chung WY, Yuan I, Desalt R, Sisti M, Bruce J, Tyoko B. MTS/p16/CDKN2 lesions in primary glioblastoma multiforme. Am J Pathol 1995;146,613-9. 22. Herman JG, Baylin SB. Gene silencing in cancer in association with promoter hypermethylation. The new England journal of medicine 2003; 349, 20042-20054. 23. Taniyama k et al. PTEN expression is maintained is sporadic colorectal tumours. J Pathol 2001; 194(3): 341-8. 24. Lee MG et al. Frequent epigenetic inactivation of RASSF1A in human bladder carcinoma. Cancer REs, 2001; 61/18):6688-92 25. Liu L et al. Frequent hypermethylation of the RASSF1A gene in prostate cancer. Oncogene 2002;21(44), 6835-40 26 Dammann R, Yang G, Pfeifer GP. Hypermethylation of the CpG island of Ras association domain family 1A (RASSF1A), a putative tumor suppressor gene from the 3p21.3 locus, occurs in a large percentage of human breast cancers. Cancer REs 2001; 61(7), 3105-9. 27. Esteller M et al. Inavtivation of the DNA repair gene MGMT and the clinical response of gliomas to alkylating agents. N Engl J Med 2000; 343:1350-4. 28. Komine C et al. Promoter hypermethylation of the DNA repair gene O6-methylguanine-DNA methyltransferase is an independent predictor of shortened progression free survival in patients with low grade diffuse astrocytoma. Brain Pathol 2003, 13:176-84. 29 Tang S et al. Relationship between alterations of p16INK4 and p14ARF genes of CDKN locus and gastric carcinogenesis. Chin Med J (Engl) 2003; 33(suppl): 1083-7. 30 Hibi K et al. Colorectal cancers with both p16 and p14 methylation show invasive characteristics. Jpn J Cancer REs 2002; 93:883-7. 31 Costello JF et al. Silencing of p16/CDKN2 expression in human gliomas by methylation and chromatin condensation. Cancer Res 1996; 56:2405-10. 32 Schmidt EE, et al. Infrequent methylation of CDKN2A(MTS1/p16) and rare mutation of both CDKN2A and CDKN2B(MTS2/p15) in primary astrocytic tumors. Br J Cancer 1997; 75:2-8. 33. Kamb A, Gruis, NA,Liu Q, Harsham K, Tavtigian SV, Stockert E, Day RS 3rd, Jonson BE, Skolnick MH. A cell cycle regulator potentially involved in genesis of many tumor types. Science. 1994, 264: 436-40 34. Nobori T, Miura K, Wu DJ, Lois A, Takabayashi K, Carson DA. Deletions of the cyclin-dependent kinase-A inhibitor gene in multiple human cancers. Nature 1994; 368:753-6). 35. Salvesen HB et al. PTEN methylation is associated with advanced stage and microsatellite instability in endometrial carcinoma. Int J Cancer 2001; 91: 22-6. 36. Kang YH, Lee HS, Kim WH. Promoter methylation and silencing of PTEN in gastric carcinoma. Lab Invest 2002; 82:285-91. 37. Soria JC et al. Lack of PTEN expression in non-small cell lung cancer could be related to promoter methylation. Clin Can REs 2002; 1178-84. 38. Baeza N et al. PTEN methylation and expression in glioblastomas. Acta Neurophatol 2003; 2:2. 39 Zysman MA, Chapman WB, Bapat B. Considerations when analyzing the methylations status of PTEN tumor suppressor gene. Am J Pathol 2002; 160:795-800.
|
|
|
|
- Cesáreo Corbacho Cuevas (11/10/2005 20:35:10)
- Javier Saez Castresana (17/10/2005 15:52:24)
- Juan Pablo Garcia de la Torre (02/11/2005 10:19:17)
|
Web mantenido y actualizado por el Servicio de informática uclm. Modificado: 16/06/2015 15:17:25
 fiogf49gjkf0d">
fiogf49gjkf0d">
 fiogf49gjkf0d">
fiogf49gjkf0d">
 fiogf49gjkf0d">
fiogf49gjkf0d">