UTILIDAD DE LA CITOGENÉTICA EN EL DIAGNÓSTICO DE LOS TUMORES LINFOIDES
Jesús María Hernández, Juan Luís García, Norma Gutiérrez, Manuel A. Sánchez, Teresa Flores1, Agustín Ríos.
Servicios de Hematología y Anatomía-Patológica1
Hospital Universitario de Salamanca
INTRODUCCIÓN
ALTERACIONES CITOGENÉTICAS DE LOS PRINCIPALES SÍNDROMES LINFOPROLIFERATIVOS.
Leucemia linfática crónica B/ Linfoma de linfocitos pequeños B
Leucemia prolinfocítica B
Inmunocitoma / Linfoma linfoplasmocítico
Linfoma de células del manto
Linfoma folicular
Linfoma MALT
Linfoma esplénico de la zona marginal
Linfoma difuso de células grandes (LDCG)
Linfoma de Burkitt
Linfomas de fenotipo T
Linfoma anaplásico de célula grande
ALTERACIONES CITOGENÉTICAS NO RELACIONADAS CON SUBTIPOS HISTOLÓGICOS CONCRETOS
REFERENCIAS
Disponible también en:
 |
INTRODUCCIÓN
Los tumores linfoides son un grupo amplio y heterogéneo de enfermedades que comparten el origen común en la proliferación clonal de células del sistema inmune, sean éstas de origen B o T. Este grupo de tumores ha sido objeto de diferentes clasificaciones, ninguna de ellas aceptada unánimemente, si bien en los últimos años la clasificación propuesta por el grupo REAL1 ha sido bien acogida por la comunidad científica. Una de las características de esta clasificación es que no sólo se basa en aspectos citomorfológicos o inmunohistoquímicos, sino también en las características citogenéticas y moleculares de los tumores linfoides.
El estudio cromosómico es una parte importante del estudio de cualquier hemopatía maligna, ya que no sólo contribuye al diagnóstico de las mismas sino que la información que aporta suele constituir un factor pronóstico de primer nivel. Estos aspectos han sido especialmente estudiados en las leucemias agudas y en los síndromes mielodisplásicos. En los tumores linfoides las alteraciones citogenéticas han sido menos analizadas; en unos casos porque, dado el bajo índice mitótico de la célula proliferante, es muy difícil la obtención de metafases analizables y, en otros -sobre todo en los linfomas agresivos- porque el estudio cromosómico en el ganglio entraña mayor dificultad que el estudio en la médula ósea (MO) y los cariotipos suelen ser muy complejos. Por todo, ello sólo recientemente se ha dispuesto de datos contrastados que permitieran valorar el papel de la citogenética en la clasificación de los tumores linfoides y que aportaran información acerca del pronóstico 2-9.
Para facilitar la comprensión de la nomenclatura citogenética es necesario recordar que la mayoría de los cromosomas humanos tienen dos brazos, uno largo (q) y otro corto (p). Las alteraciones que pueden encontrarse son de dos clases: primarias, cuando están ligadas específicamente a cada tipo de tumor, y secundarias, cuando se añaden a las anteriores. Dos son también los tipos de alteraciones: numéricas y estructurales. En la tabla I se ofrece un glosario con la nomenclatura de las alteraciones cromosómicas. La valoración de una clona anormal exige analizar e interpretar un número suficiente de metafases. Los cromosomas y sus alteraciones se identifican de acuerdo con las sucesivas recomendaciones del Sistema Internacional para la Nomenclatura de la Citogenética Humana (ISNC, 1995)11. Se considera una alteración clonal cuando se detectan dos o más metafases con la misma anomalía estructural o con ganancia de algún cromosoma, pero si es una pérdida ha de aparecer en al menos 3 mitosis.
En los últimos años se han desarrollado una serie de técnicas complementarias de la citogenética convencional. Por ello, en la moderna citogenética hay que incluir los resultados obtenidos de combinar estos estudios con la hibridación in situ12-15, con la hibridación genómica comparada (HGC)16 y, en un futuro próximo, con los de la hibridación "in situ" multicolor (cariotipaje espectral en colores17, bandeo multicolor, etc).
Tabla I. Glosario de la nomenclatura citogenética
| Tipo de alteración | Nombre | Abreviatura | Descripción |
| Trisomía | + | Ganancia de un cromosoma | |
| Monosomía | - | Pérdida de un cromosoma | |
| Numéricas | |||
Hiperdiploidía |
Número de cromosomas > 46 | ||
| Hipodiploidía | Número de cromosomas < 46 | ||
| Deleción | del | Pérdida de un segmento cromosómico | |
| Inversión | inv | Giro de 180 ºC del material dentro del mismo cromosoma | |
| Isocromosoma | i | Duplicación de todo el brazo de un cromosoma con pérdida del otro brazo | |
Estructurales |
|||
| Traslocación | t | Intercambio recíproco de material cromosómico entre 2 o más cromosomas | |
| Derivativo | der | Intercambio no recíproco entre dos cromosomas |
ALTERACIONES CITOGENÉTICAS DE LOS PRINCIPALES SÍNDROMES LINFOPROLIFERATIVOS
Leucemia linfática crónica B/ Linfoma de linfocitos pequeños B
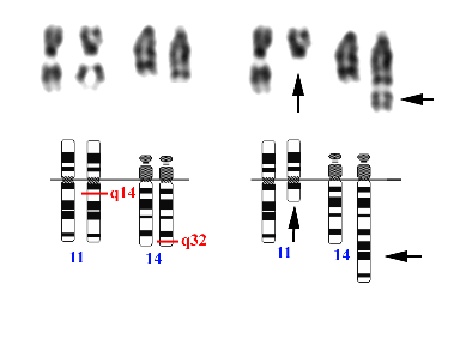
Se han publicado casi 3000 casos de LLC con estudios citogenéticos. El 37% de los enfermos presentaba alteraciones clonales y la alteración más frecuente es la trisomía del cromosoma 12 (+12), observada en el 13% de todos los casos con LLC, y las aberraciones estructurales (especialmente deleciones) de los cromosomas 13q (7%), 11q (7%) (Fig. 1) y 6q (7%). Otras alteraciones numéricas -como la +3 o la +18- o estructurales -en el cromosoma 14, 17 o en el 7- son menos habituales 2,5,13,18-25 (tabla II). Estos resultados han sido confirmados por estudios de HIS. La mayoría de ellos han realizado el estudio de la trisomía 12 mediante el uso de una sonda pericentromérica específica de este cromosoma, que permite detectar trisomía 12 en el 22% de los más de 1500 enfermos con LLC estudiados 13-15,19,21,22,24,26-29.
 |
| Figura 1. Cariotipo parcial y representación esquemática de una delección del brazo largo del cromosoma 11 observada en un enfermo con LLC. |
Además, se ha demostrado que la trisomía 12 aparece con mayor frecuencia en las LLC que presentan una morfología atípica o tienen expresión fuerte de smIg y/o FMC7(+). Los estudios de HIS y de técnicas moleculares, con sondas específicas de locus o con cromosomas artificiales de levaduras (YACs), han servido también para delimitar las regiones genómicas que se encuentran delecionadas en las proliferaciones linfoides. Así, se ha demostrado que las LLC con deleción del cromosoma 13q suelen tener perdido el gen del retinoblastoma (RB1), pero que este gen no está implicado en todas las LLC30, sino otro situado teloméricamente al RB1. La región comúnmente delecionada en la del(11q) se sitúa entre 11q22.3 y 11q23.1, aunque aún no se ha identificado el gen que se pierde en esta deleción31. Además de estas alteraciones, en la LLC pueden encontrarse cambios típicos de otras proliferaciones linfoides; así, en algunas LLC se observa la t(11;14)32 o, con menor frecuencia, la t(14;18)5. Por lo que respecta al valor pronóstico, la LLC con 13q- o cariotipo normal suele asociarse con buena evolución; por el contrario, los enfermos con 11q-, 17p- o cariotipo complejo tienen peor pronóstico. La trisomía 12 presenta un pronóstico intermedio.
Los estudios de hibridación genómica (HGC) comparada han corroborado los resultados citogenéticos y de HISF. Los cambios observados en la LLC son la trisomía 12, 11q-, 17p- y 13q- 33,34.
Esta rara enfermedad suele presentar cambios citogenéticos en la mayoría de los casos. La alteración más frecuente (50% de los enfermos) es el 14q+ (es decir, la aparición de un fragmento de material cromosómico unido al cromosoma 14); la mayoría de estos enfermos con 14q+ suelen tener una t(11;14), similar a la que se observa en el linfoma del manto. Otras alteraciones presentes en la LPL son la +12, 1q- y 6q- 35.
Tabla II. Alteraciones citogenéticas y oncogenes implicados en los LNH
| Histología | Cambio cromosómico | Oncogén/es |
LNH-B o T |
1p / 1q |
? / ? |
LCGD-B |
t(2;3)(q12;q27) |
IGK/BCL-6 |
LNH CD-30+ |
t(2;5)(p23;q35) |
ALK/NPM |
t(2;8)(p12;q24) |
IGK/C-MYC |
|
LCGD-B |
t(3;14)(q27;q32) |
BCL-6/IGH |
LCGD-B |
t(3;22)(q27;q11) |
BCL-6/IGL |
+3 |
? / ? |
|
LNH-B |
del(6q) |
? |
LNH-B |
+7 |
? |
7p15 |
TCR- d |
|
LEV/LZME |
del(7q) |
? |
7q35 |
TCR- b |
|
t(8;14)(q24;q32) |
C-MYC/IGH |
|
t(8;22)(q24;q11) |
C-MYC/IGL |
|
INMUNOCITOMA |
t(9;14)(p13;q32) |
PAX5 /IGH |
LNH-B |
t(10;14)(q24;q32) |
LYT10/IGH |
t(11;14)(p13;q11) |
RBTN2/TCR a/d |
|
t(11;14)(q13;q32) |
BCL-1/IGH |
|
t(11;18)(q21;q21) |
? / ? |
|
LLBDD/LCGD-B |
+12? |
|
t(14;18)(q32;q21) |
IGH/BCL-2 |
|
14q11 |
TCR a/TCRd |
|
t(18;22)(q21;q11) |
BCL-2/IGL |
________________________________________________________________________
LCGD-B: linfoma de células grandes difuso B. LB: linfoma de Burkitt. LF: linfoma folicular
MALT: linfoma asociado a mucosas. LEV/LZME: linfoma esplénico con linfocitos vellosos/ linfoma de la zona marginal esplénica. LM: linfoma del manto. LLBDD: linfoma linfocítico bien diferenciado difuso.
Inmunocitoma / Linfoma linfoplasmocítico
Hay muy pocos datos acerca de las alteraciones citogenéticas de este grupo de linfomas. Sin embargo, se ha descrito que la t(9;14)(p13;q32) donde el gen PAX-5 se trasloca a la región de las cadenas pesadas de las inmunoglobulinas, aparece en el 50% de los enfermos con inmunocitoma 36-37.
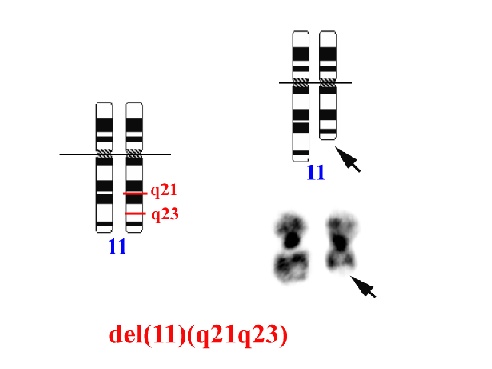
La alteración citogenética característica del linfoma de células del manto (LM) y que aparece en más de la mitad de los enfermos con LM es la t(11;14)(q13;q32) 5,35,38 (Fig. 2). En ella el gen BCL-1/PRAD1, que codifica para la Ciclina D-1 (implicada en la regulación del ciclo celular), se trasloca a la región del gen de la cadena pesada de las inmunoglobulinas (tabla III). Esta alteración no es exclusiva de este tipo de linfomas y puede observarse también en otras proliferaciones linfoides (LLC, mieloma múltiple, linfomas marginales32,39). Mediante HISF es posible observar la t(11;14) en más del 90% de los LM 40,41.
Por el contrario, mediante Southern o PCR sólo se detecta la traslocación en el 65% de los LM debido a que el gen BCL-1/PRAD1 es muy grande y para su estudio se requiere la digestión con distintos enzimas y varias sondas para su hibridación. En el 20-40% de los LM se observan otras alteraciones asociadas a la t(11;14). Las más frecuentes afectan al cromosoma 1 o a 6q 5,38. Se desconoce el significado pronóstico de las mismas, aunque se han relacionado con evolución clonal en los LM. En las variantes blásticas de LM los cambios citogenéticos suelen afectar a varios cromosomas, dando lugar a cariotipos complejos o hiperdiploides.
Los hallazgos detectados con mayor frecuencia mediante HGC en el LM son 3q+, 13q-, 1p-, 6q- y 8q+. Además, se han descrito amplificaciones en varias regiones genómicas (3p, 8q y 13q) 42.
Tabla III. BCL y proliferaciones linfoides B
Tipo de linfoma |
Citogenética |
BCL |
Gen traslocado |
t(11;14)(q13;q32) |
1 |
IgH |
|
t(14;18)(q32;q21) |
2 |
IgH, Igk,Igl |
|
t(14;19)(q32;q13) |
3 |
IgH |
|
Célula grande |
reorden. 3q27 |
6 |
IgH, Igk,Igl y otros |
Líneas celulares (LB) |
12q21 |
7 |
|
Linfomas agresivos |
15q11-13 |
8 |
|
Linfomas agresivos |
t(1;14)(q21;q32) |
9 |
IgH |
Los linfomas foliculares (LF) presentan, de manera característica, en más del 70% de los casos la t(14;18)(q32;q21) 9,43 en la cual el gen BCL-2 (situado en 18q21) se trasloca a la región 14q32. La disrregulación del gen BCL-2, implicado en mecanismos de apoptosis, prolonga la supervivencia de las células linfomatosas (tablas II y III). Al igual que ocurre en el LM, la t(14;18) o sus variantes la t(2;18) o la t(18;22) no son exclusivas del LF sino que pueden aparecer hasta en el 25% de los linfomas difusos de células grandes (confiriéndoles mal pronóstico) y en la LLC. En más de la tercera parte de los LF la t(14;18) se asocia con alteraciones en 1q, con 6q- y con 17p-. La aparición de estas anomalías asociadas se relaciona con evolución tumoral y ensombrece el pronóstico de los LF 44,45. Mediante HGC se observan Xp+, +7 6q- 46.
La alteración más frecuente es la trisomía 3, hallazgo muy común en otros grupos de LNH 47,48 y que ha sido confirmado por HISF 49. Sin embargo, es más específica de este tipo de linfomas la t(11;18)(q21;q21)50 en la que aún se desconoce qué genes están implicados, pero que se asocia de manera característica a MALT de bajo grado 51 (tabla II). Además de estas dos alteraciones se han descrito otros cambios, comunes a otros LNH, como las alteraciones en el cromosoma 1, +7, +12 y +18 48. Mediante HGC se han confirmado la mayoría de estas alteraciones. Se han observado con mayor frecuencia 3q+, +18,Xp+ y 17p-. Además, algunos pacientes presentan amplificaciones en la región más telomérica de 18q 52.
Linfoma esplénico de la zona marginal
A diferencia de lo observado en los LM, LF y MALT, los linfomas esplénicos de la zona marginal (LEZM) no tienen ninguna alteración citogenética específica, si bien la deleción del brazo largo del cromosoma 7 se ha detectado con mayor frecuencia que en otros linfomas 5,39(Fig. 3). Otros cromosomas alterados son el 1, el 3 y el 8; pero la aparición de trisomía 3 es menos frecuente que en los linfomas marginales. Mediante pintado cromosómico se ha confirmado que los cromosomas 1, 3, 7 y 8 están frecuentemente alterados en el LEZM 53. Además, los estudios de HISF con YACs han permitido delimitar la región común delecionada en 7q en el límite entre 7q31 y 7q32 54.
Linfoma difuso de células grandes (LDCG)
Los LDCG representan un grupo muy heterogéneo de LNH-B. Por ello, las alteraciones citogenéticas, aunque recurrentes, son muy diversas. La mayoría de los LDCG presentan alteraciones citogenéticas y sus cariotipos suelen ser complejos 55.
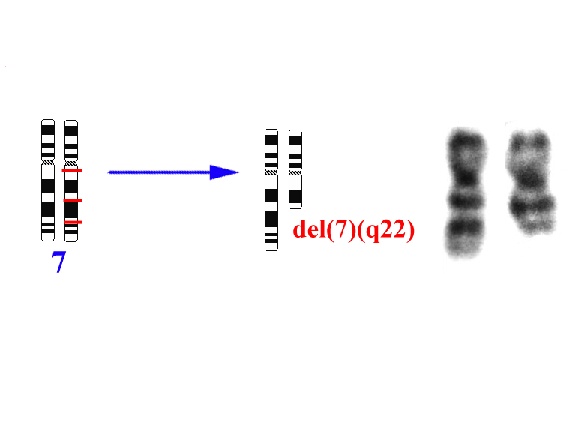 |
| Figura 3. Ideograma y cariotipo parcial de un enfermo con LEZM y delección del brazo largo del cromosoma 7 |
Las anomalías estructurales observadas con mayor frecuencia afectan a 14q32, 18q21, 1q, 8q y 6q; entre los cambios numéricos se observan trisomías de los cromosomas 7 y 12 55-57. Los enfermos con SIDA y LCGD-B muestran alteraciones similares al resto de los enfermos, aunque se ha observado una mayor frecuencia de 6q- 58. Todas estas alteraciones condicionan el pronóstico de los LCGD-B. Se asocian con una menor supervivencia la existencia de cariotipos complejos o las alteraciones en 1q, 6q, 1p, +7 o +12 9-45.
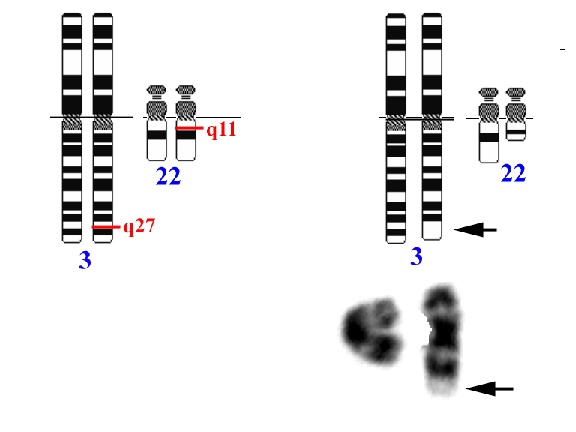
Una de las regiones cromosómicas relacionada con los LCGD-B se sitúa en 3q27 y es difícil de analizar por citogenética convencional (Fig. 4). Sin embargo, mediante estudios de HIS y de biología molecular se ha demostrado que 3q27 está alterada en la cuarta parte de los enfermos con LCGD-B 57-59. En esta región se sitúa el gen BCL-6, una de cuyas funciones es la de controlar el centro germinal y que puede traslocarse con diferentes cromosomas, aunque con mayor frecuencia se reordena con 14q32 y 22q11 (tabla III). El valor pronóstico del reordenamiento de BCL-6 en los LCGD-B es aún controvertido. Para algunos se asociaría con una mayor supervivencia 59, pero otros no han confirmado estos hallazgos 57. Las alteraciones de BCL-6 no son exclusivas de los LCGD-B y también se han descrito en los linfomas marginales y en los linfomas del manto 60. De igual manera, los LCGD-B pueden presentar otras alteraciones moleculares, como reordenamientos de BCL-2, C-MYC, REL, P16, P53, etc 61.
El estudio de los LCGD-B mediante hibridación genómica comparada (HGC) ha corroborado los hallazgos citogenéticos, confirmando que la mayoría de estos linfomas presentan, como alteraciones cromosómicas más frecuentes, +X, +1q, 6q-, +7 y +3 62-64. En algunos casos se han descrito amplificaciones en 18q21, 2p y 6p 62-65.
El linfoma de Burkitt (LB) fue el primer modelo de cáncer humano estudiado mediante técnicas moleculares. Las alteraciones citogenéticas y moleculares del LB son idénticas a las que se observan en la LAL-3 y afectan a la región 8q24.1, donde se localiza el oncogén C-MYC. Hay tres tipos de traslocaciones: la t(8;14)(q14;q32) es la más frecuente (65%-80%) y en ella el oncogén C-MYC se reordena con el gen de la cadena pesada de las inmunoglobulinas. Las traslocaciones variantes son la t(8;22)(q24;q11) -observada en el 11%-15% de los casos y en la que C-MYC se reordena con el gen de la cadena ligera l de las inmunoglobulinas- y la t(2;8) (p12;q24) -observada en el 8% de los pacientes y en la que C-MYC se trasloca con el gen de la cadena ligera k (tabla II)-. En las dos traslocaciones variantes el gen C-MYC permanece en el cromosoma 8, mientras que en la t(8;14) C-MYC se trasloca a 14q32. En el LB asociado a infección por el VIH las alteraciones citogenéticas y moleculares son superponibles a las encontradas en las forma esporádica del LB 66.
Cualquiera de las tres traslocaciones que aparecen en el LB suelen ir asociadas con otras en el 70% de los enfermos. Las más frecuentes son las duplicaciones parciales del brazo largo del cromosoma 1 (1q12-31), reordenamientos de 13q y 17q, trisomías de los cromosomas 7 y 12, y 6q-. En un 15% de los LB no se detectan alteraciones citogenéticas, aunque todos los casos tienen reordenado el oncogén C-MYC. Aunque en los enfermos con LB se ha sugerido la existencia de una asociación entre la t(8;14) y un mejor pronóstico, este hallazgo se ha establecido en base a estudios retrospectivos y, por tanto, necesita ser confirmado 67.
Bajo la denominación de linfomas T se agrupa un grupo heterogéneo de neoplasias cuya clasificación ha sido siempre controvertida. Este hecho, unido a la baja prevalencia de estas enfermedades (suponen menos del 15% de todos los LNH) explica la carencia de estudios citogenéticos sistematizados en los LNH-T. Las neoplasias de estirpe T suelen presentar alteraciones citogenéticas en las regiones cromosómicas en las que se sitúan los genes de los receptores de la Célula T (RCT). En 14q11 están los RCT a y d; en 7q35 está el RCT-b y en 7p15 está el RCT-g (tabla II). Curiosamente, algunas neoplasias T presentan de manera recurrente reordenamientos de la región 14q32, donde se sitúa el gen JH y otro gen implicado en estos procesos, denominado TCL-1 .
Más del 90% de los enfermos con leucemia-linfoma T del adulto presentan alteraciones citogenéticas. Las más frecuentes se localizan en el cromosoma 14q y son la t(14;14)(q11;q32), la inv(14) y el 14q-. En esta enfermedad se han descrito reordenamientos de 14q con otros cromosomas (1, 3q, 10p y 12q). Otras alteraciones son el 6q- y trisomías de los cromosomas 3, 7 y 21 68 (Tabla IV).
Se conoce muy poco acerca de las anomalías cariotípicas presentes en la micosis fungoide y en el síndrome de Sezary. Los cariotipos suelen ser complejos y, curiosamente, la afectación de las regiones donde se localizan los RCT es infrecuente. Sin embargo, pueden encontrarse 1p-, 6q- y reordenamientos de 14q32, 10q y 17q 69.
Tabla IV. Cambios
cromosómicos en los LNH-T
__________________________________________
t(14;V)(q11;V)1
inv(14)(q11q32)
t(7;7)(p15;q11)
t(7;V)(q34;V)2
inv(7)(p14q35)
del(9)(p13-22)
t(9;17)(q34;q23)
t(11;14)(p13;q11)
____________________________________________
1
V: cromosoma 1, 9, 10, 11, 14 o 192
V: cromosoma 1, 7, 8, 10, 11, 14
Linfoma anaplásico de célula grande
Este grupo de linfomas, caracterizado por la expresión del CD30, se asocia de manera característica con la t(2;5)(p23;q35) en la que se reordenan los genes ALK, que es una kinasa, con el gen de la proteína nucleolar NPM (localizado en 5q35) 70. La t(2;5) es difícil de observar por citogenética convencional, por lo que para ponerla de manifiesto es conveniente realizar estudios de HIS o de biología molecular 71.
Alteraciones citogenéticas no relacionadas con subtipos histológicos concretos
Algunas anomalías cromosómicas aparecen de manera recurrente en los LNH, aunque no específicamente asociadas con un subtipo histológico. Se han descrito todo tipo de alteraciones, numéricas y estructurales, que afectan a cualquier cromosoma. Las más frecuentes son las alteraciones estructurales de 1p, 1q, 3q y 9q; las deleciones del brazo largo del cromosoma 6; y las trisomías de los cromosomas 3, 7 y 18 9,45.
El valor pronóstico de las alteraciones citogenéticas en los LNH debe ser analizado en estudios prospectivos, que incluyan un gran número de enfermos uniformemente tratados. Algunos estudios preliminares sugieren que la +7, +18 o 6q- son cambios secundarios que implican una evolución clonal y, a veces, histológica de los LNH. Por ello, estas alteraciones suelen conllevar peor pronóstico. Por el contrario, la trisomía del cromosoma 3 se ha relacionado con una mejor respuesta a la quimioterapia 9,45,73.
A modo de resumen, se puede afirmar que en los LNH los estudios citogenéticos, de HISF y de hibridación genómica comparada son herramientas de suma utilidad, ya que ayudan a clasificar las proliferaciones linfoides y han demostrado su relación con el pronóstico de estas enfermedades.
- Harris NL, Jaffe ES, Stein H, Banks PM, Chan JKC, Cleary ML et al. A revised European-American classification of lymphoid neoplasms: a proposal from the international lymphoma study group. Blood 1994;84:1361-1392
- Juliusson G, Oscier DG, Fitchett M, Ross F, Stockdill G, Mackie MJ et al. Prognostic subgroups in B-cell chronic lymphocytic leukemia defined by specific chromosomal abnormalities. New Engl J Med 1990;323;720-724.
- Juliusson G, Gahrton G. Cytogenetics in CLL and related disorders. Baillieres Clin Haematol 1993;6;821-848.
- Asou H, Takechi M, Tanaka K, Tashiro S, Dohy H, Ohno R et al. Japanese B cell chronic lymphocytic leukaemia: a cytogenetic and molecular biological study. Br J Haematol 1993;85:492-497.
- Hernández JM, Mecucci C,Criel A, Meeus P, Michaux L, Van Hoof A et al. Cytogenetic analysis of B-cell chronic lymphoid leukemias classified according to morphologic and immunophenotypic (FAB) criteria. Leukemia 1995; 9: 2140-2146.
- Solé F, Woessner S, Pérez-Losada A, Florensa L, Montero S, Asensio A et al. Cytogenetic studies in 76 cases of B chronic lymphoproliferative disorders. Cancer Genet Cytogenet 1997; 93: 160-166.
- Kluin-Nelemans HC, Beverstock GC, Mollevanger P, Wessels H, Hoogendoorn E, Willemze R, Falkenburg JH. Proliferation and cytogenetic analysis of hairy cell leukemia upon stimulation via the CD40 antigen. Blood 1994;84:3134-3141.
- Haglund V, Juliusson G, Stellan B, Gahrton G. Hairy cell leukemia is charaterized by clonal chromosome abnormalities clustered to specific regions. Blood 1994;83:2637-2645.
- Offit K, Jhanvar SC, Ladanyi M, Filippa DA, Chaganti RSK. Cytogenetic analysis of 434 consecutively ascertained specimens of non-Hodgkin's lymphoma: correlations between recurrent aberrations, histology, and exposure to cytotoxic treatment. Genes Chrom Cancer 1991;3:189-201.
- García JL, Hernández JM, Gutiérrez NC, Fernández P, Ríos A. La citogenética en el estudio de las hemopatías malignas. Sangre 1996; 41: 289-296.
- ISCN. Guidelines for Cancer Cytogenetics. Supplement to An International System for Human Genetics Nomenclature, Mitelman F, ed. Basel: S. Karger, 1995.
- Hernández JM, Tabernero MD, García JL. Aplicaciones de la hibridación in situ fluorescente al estudio de las neoplasias hematológicas. Sangre 1996; 41: 305-310.
- Escudier SM, Pereira-Leahy JM, Drach JW, Weier HU, Goodacre AM, Cork MA, et al. Fluorescent in situ hybridization and cytogenetic studies of trisomy 12 in chronic lymphocytic leukemia. Blood 1993;81:2702-2707.
- Tabernero MD, San Miguel JF, García JL, García-Isidoro M, Wiegant J, Ciudad J et al. Clinical, biological and immunophenotypical characteristics of B-cell chronic lymphocytic leukemia with trisomy 12 by fluorescence in situ hybridization. Cytometry 1995; 22: 217-222.
- Que TH, García Marco J, Ellis J, Matutes E, Brito Babapulle V, Catovsky D. Trisomy 12 in chronic lymphocytic leukemia detected by fluorescence in situ hybridization: analysis by stage, immunophenotype, and morphology. Blood 1993;82:571-575.
- Bentz M, Huck K, du Manoir S, Joos S, Werner CA, Fischer K, Döhner h, Lichter P. Comparative genomic hybridization in chronic B-cell leukemias shows a high incidence of chromosomal gains and losse. Blood 1995; 85: 3610-3618.
- Veldman T, Vignon C, Schrock E, et al. Hidden chromosomal abnormalities in haematological malignancies detected by multicolor spectral karyotyping. Nat Genet 1997; 15: 406-410.
- Matutes E, Oscier D, García Marco J, et al. Trisomy 12 defines a groupof CLL with atypical morphology: correlation between cytogenetic, clinical and laboratory features in 544 patients. Br J Haematol 1996;92: 382-8.
- García-Marco J, Price CM, Ellis J, et al. Correlation of trisomy 12 with proliferating cells by combined immunocytochemistry and fluorescence in situ hybridization in chronic lymphocytic leukaemia. Leukemia 1996; 1996; 10: 1705-1711.
- Philip P, Geisler C, Hansen MM, et al. Aberrations of chromosome 6 in 193 newly diagnosed untreated cases of chronis lymphocytic leukemia. Cancer Genet Cytogenet 1991; 53:35-43.
- Coignet L, Bertheas MF, Vasselon C, et al. Trisomy 12 in chronic lymphocytic leukemia: Interphase study by in situ hybridization in 75 patients. Nouv Rev Fr Hematol 1993; 35: 129-134.
- Woessner S, Solé F, Pérez-Losada A, et al. Trisomy 12 is a rare cytogenetic finding in typical lymphocytic leukemia. Leuk Res 1996; 20: 369-374.
- Delhomme-Bachy M, Bertheas MF, Vasselon C, et al. Chromosome studies in stimulated lymphocytes of 50 patients with B-cell chronic lymphocytic leukemia. Nouv Rev Fr Hematol 1992; 34: 175-182.
- Döhner H, Pilz T, Fisher k et al. Molecular cytogenetic analysis of RB-1 deletions in chronic B-cell leukemias. Leuk Lymph 1994; 16: 97-103.
- Finn WG, Thangavelu M, Yelavarthi KK, et al. Karyotype correlations with peripheral blood morphology and immunophenotype in chronic lymphocytic leukemia. Am J Clin Pathol 1996; 105: 458-467.
- Witzig TE, Borell TJ, Herarth JF, et al. Detection of trisomy 12 by FISH in untreated B-chronic lymphocytic leukemia: Correlation with stage and CD20 antigen expression intensity. Leuk Lymph 1994; 14: 447-451.
- Cuneo A, Bigoni R, Balboni M, et al. Trisomy 12 in chronic lymphocytic leukemia and hairy cell leukemia: A cytogenetic and interphase study. Leuk Lymph 1994; 167-172.
- Raghoebier S, Kibelaar RE, Kleiverda JK, et al. Mosaicism of trisomy 12 in chronic lymphocytic leukemia detected by non-radiactive in situ hybridization. Leukemia 1992; 6: 1220-1226.
- Bienz N, Min C, Watters JJ, et al. Comparison of conventional karyotyping and direct fluorescence in situ hybridization for detection of trisomy 12 in B-CLL. Br J Haematol 1994 (suppl); 86: 75.
- Liu Y, Szekely L, Grander D, Söderhäll S, Juliusson G, Gahrton G, Linder S, Einhorn S. Chronic lymphocytis leukemia cells with allelic deletions at 13q14 commonly have one intact RB1 gene: evidence for a role of an adjacent locus. Proc Natl Acad Sci USA 1993; 90:8697-8701.
- Stilgenbauer S, Liebisch P, James MR et al. Molecular cytogenetic delineation of a novel critical genomic region in chromosome bands 11q22.3-q23.1 in lymphoproliferative disorders. Proc Natl Acad Sci 1996; 93: 11837-11841.
- Cuneo A, Balboni M, Piva N, Rigolin M, Roberti MG, Mejak C et al. Atypical chronic lymphocytic leukaemia with the t(11;14)(q13;q32): karyotype evolution and prolymphocytic transformation. Br J Haematol 1995;90: 409-416.
- Bentz M, Huck K, du Manoir S, et al. Comparative genomic hybridization in chronic B-cell leukemias shows a high incidence of chromosomal gains and losses. Blood 1995; 85: 3610-3618.
- Karhu R, Knuutila S, Kallioniemi OP, et al. Frequent loss of the 11q14-24 region in chronic lymphocytic leukemia: a study by comparative genomic hybridization. Genes Chromosom Cancer 1997; 19: 286-290.
- Brito-Babapulle V, Ellis J, Matutes E, Oscier D, Khokhar T, MacLennan K, Catovsky D. Translocation t(11;14)(q13;q32) in chronic lymphoid disorders. Genes Chromosom Cancer 1992;5:158-165.
- Offit K, Parsa NZ, Filippa D, et al. t(9;14)(p13;q32) denotes a subset of low-grade non-Hodgkin’s lymphoma with plasmacytoid differentiation. Blood 1992; 80: 2594-2599.
- Iida S, Rao PH, Nallasivam P, et al. The t(9;14)(p13;q32) chromosomal translocation associates with lymphoplasmacytoid lymphoma involves the PAX-5 gene. Blood 1996; 88: 4110-4117.
- Vandenberghe E, de Wolf-Peeters C, van den Oord J, et al. Translocation t(11;14)(q13;q32): a cytogenetic abnormality associated with B-cell lymphomas of non-follicle centre cell lineage. J Pathol 1991; 163: 13-18.
- Oscier DG, Matutes E, Gardiner A, et al. Cytogenetic studies in splenis lymphoma with villous lymphocytes. Br J Haematol 1993; 85: 487-491.
- Cuneo A, Bigoni R, Negrini M, et al. Cytogenetic and interphase cytogenetic characterization of atypical chronic lymphocytic leukemia carrying BCL1 trasnlocation. Cancer Res 1997; 57: 1144-1150.
- Monteil M, Callanan M, Dascalescu C, et al. Molecular diagnosis of t(11;14) in mantle cell lymphoma using two-colour interphase floureswcence in situ hybridization. Br J Haematol 1996; 93: 656-660.
- Monni O, Oinonen R, Elonen E, et al. Gain of 3q and deletion of 11q22 are frequent aberrations in mantle cell lymphoma. Genes Chromosom Cancer 1998; 21: 298-307.
- Schouten HC, Sanger WG, Weisemburger DD, Anderson J, Armitage JO. Chromosomal abnormalities in untreated patients with non-Hodgkin's lymphoma: associations with histology, clinical characteristics, and treatment outcome. Blood 1990;75:1841-1847.
- Tilly H, Rossi A, Stamatoullas A, et al. Prognostic value of chromosomal abnormalities in follicular lymphoma. Blood 1994; 84: 1043-1049.
- Pirc-Danoewinata H, Chott A, Onderka E, Drach J, Schlögl E, Jäger U et al. Karyotype and prognosis in non- Hodgkin's lymphoma. Leukemia 1994;8:1929-1939.
- 46.Bentz M, Werner CA, Döhner H, et al. High incidence of chromosomal imbalances and gene amplifications in the classical follicular variant of follicle center lymphoma. Blood 1996; 88: 1437-1444.
- Robledo M, Benitez J, Rivas C, et al. Cytogenetic study of B-cell lymphoma of mucosa-associated lymphoid tissue. Cancer Gente Cytogenet 1992; 62: 208-209.
- Dierlamm J. Pittaluga S, Wlodarska I, et al. Marginal zone B-cell lymphomas of different sites share similar cytogenetic and morphological features. Blood 1996; 87: 299-307.
- Dierlamm J, Michaux L, Wlodarska I, et al. Trisomy 3 in marginal zone B-cell lymphoma: a study based on cytogentic analysis and fluorescence in situ hybridization. Br J Haematol 1996; 93: 242-249.
- Horsman D, Gascoyne R, Klassa R, Coupland R. t(11;18)(q21;q21.1): a recurring translocation in lymphomas of mucosa-associated lymphoid tissue (MALT)?. Genes Chromosomes Cancer 1992; 4: 183-187.
- Ott G, Kartzemberg T, Greiner A, et al. The t(11;18)(q21;q21) chromosome translocation is a frequent an specific aberration in low-grade but not high-grade malignant non-Hodgkin’s lymphomas of the mucosa-associated lymphoid tissue (MALT) type. Cancer Res 1997; 57: 3944-3948.
- Dierlamm J, Rosenberg C, Stul M, et al. Characteristic pattern of chromosomal gains and losses in marginal zone B cell lymphoma detected by comparative genomic hybridization. Leukemia 1997; 11: 747-758.
- Solé F, Woessner S, Florensa L, et al. Frequent involvement of chromosomes 1, 3, 7 and 8 in splenic marginal zone B-cell lymphoma. Br J Haematol 1997; 98: 446-449.
- Hernández JM, Schoenmakers EFPM, Dal Cin P, Michaux L, Van de Ven WJM, Van den Berghe H. Molecular delineation of the commonly deleted segment in mature B-cell lymphoid neoplasias with deletion of 7q. Genes Chrom Cancer 1997; 18: 147-150.
- Koduru PRK, Filippa DA, Richardson ME et al. Cytogenetic and histologic correlations in malignant lymphoma. Blood 1987; 69. 97-102.
- Pastore C, Carbone A, Gloghini A, et al. Association of 6q deletions with AIDS-related diffuse large cell lymphoma. Leukemia 1996; 10: 1051-1053.
- Bastard C, Deweindt C, Kerckaert JP, et al. LAZ3 rearrangements in non-Hodgkin?s lymphoma: correlation with histology, immnuophenotype, karyotype, and clinical outcome in 217 patients. Blood 1994; 83: 2423-2427.
- Offit K, Lo Coco F, Louie Dc, et al. Rearrangement of the BCL-6 gene as aprognostic marker in diffuse large-cell lymphoma. N Engl J Med 1994; 331: 74-80.
- Wlodarska I, Mecucci C, Stul M, et al. Fluorescence in situ hybridization identifies new chromosomal changes involving 3q27 in non-Hodgkin?s lymphomas with BCL6/LAZ3 rearrangement. Genes Chromosom Cancer 1995; 14: 1-7.
- Dierlamm J, Pittaluga S, Stul M, et al. BCL-6 rearrangements also occur in marginal zone lymphoma. Br J Haematol 1998; 98: 719-725.
- Volpe G, Vitolo U, Carbone A et al. Molecular heterogeneity of B-lineage diffuse large cell lymphoma. Genes Chromosom Cancer 1996; 16: 21-30.
- Monni O, Joensuu H, Franssila K, Knuutila S. DNA copy number changes in diffuse large B-cell lymphoma - Comparative genomic hybridization study. Blood 1996; 87: 5269-5278.
- Werner CA, Döhner H, Joos S, et al. Hish-level DNA amplifications are common genetic aberrations in B-cell neoplasms. Am J Pathol 1997; 151: 335-342.
- Knuutila S, Björkqvist AM, Autio K, et al. DNA copy number amplifications in human neoplasms. Am J Pathol 1998; 152: 1107-1123.
- Houldsworth J, Mathew S, Rao PH, et al. REL proto-oncogene is frequently amplified in extranodal diffuse large cell lymphoma. Blood 1996; 87: 25-29.
- Mitelman F, Mertens F, Johansson B. A breakpoint map of recurrent chromosomal rearrangements in human neoplasia. Nature Genet 1997; 417-474.
- Kornblau SM, Goodacre A, Cabanillas F. Chromosomal abnormalities in adult non-endemic Burkitt?s lymphoma and leukemia: 22 new reports and a review of 148 cases from the literature. Hematol Oncol 1991; 9: 63-78.
- Schlegelberger B, Himmler A, Bartless H, et al. Recurrent chromosome abnormalities in peripheral T-cell lymphomas. Cancer Genet Cytogenet 1994; 78: 15-22.
- Thangavelu M, Finn WG, Yelavarthi KK, et al. Recurring structural chromosome abnormalities in peripheral blood lymphocytes of patients with mycosis fungoides/Sezary syndrome. Blood 1997; 89: 3371-3377.
- Lamant L, Meggeto F, Al Saati T, et al. High incidence of the t(2;5)(p23;q35) translocation in anaplastic large cell lymphoma and its lack of detection in Hodgkin?s disease. Comparison of cytogenetic analysis, reverse transcriptase-polymerase chain reaction, and P-80 immunostaining. Blood 1996; 87: 284-291.
- Mason DY, Bastard C, Rimokh R, et al. CD30-positive large cell lymphomas (Ki-1 lymphoma) are associated with a chromosomal translocation involving 5q35. Br J Haematol 1990; 74: 161-168.
- Bloomfield CD, Arthur DC, Frizzera G, Levine EG, Peterson BA, Gajl-Peczalska J. Nonrandom chromosome abnormalities in lymphoma. Cancer Res 1983;43:2975-2984.
- Levine EG, Arthur DC, Frizzera G et al. Cytogenetic abnormalities predict clinical outcome in non-Hodgkin lymphoma. Ann Int Med 1988; 108: 14-20.