

|
Perfil genético de los tumores embrionarios del sistema nervioso María del Mar Inda*, Teresa Tuñón**, Javier S. Castresana* |
|
|
Los tumores embrionarios son tumores indiferenciados
de células redondas que muestran diferentes patrones de diferenciación.
Representan un grupo heterogéneo de tumores del sistema nervioso
central (SNC) y del periférico (SNP), y tanto su origen como su
clasificación histopatológica no están claras. El meduloblastoma es el
PNET del SNC más común en la edad pediátrica, suponiendo el 20-25% de
los tumores cerebrales. Los PNET supratentoriales son histológicamente
similares a los meduloblastomas, pero su pronóstico es más agresivo que
el de éstos. El tumor embrionario más frecuente del SNP es el
neuroblastoma, que presenta características histológicas similares a
los PNET centrales con diferenciación neuronal. A fin de estudiar las posibles diferencias en el perfil genético de los tumores embrionarios del sistema nervioso, realizamos técnicas de análisis de deleciones homocigóticas, hipermetilación de promotores, y expresión a nivel de RNA en diversos genes supresores de tumores, así como la técnica de hibridación genómica comparada (CGH) sobre un total de 78 tumores embrionarios, que incluian 52 PNET del SNC (37 meduloblastomas y 15 PNET supratentoriales), y 26 tumores neuroblásticos del SNP (16 neuroblastomas, 7 ganglioneuroblastomas, y 3 ganglioneuromas). Los resultados confirman la hipótesis de que los tumores embrionarios del sistema nervioso constituyen diferentes entidades genéticas con diferentes alteraciones moleculares, independientemente de la apariencia histológica similar que presentan.
|
||
|
|
Los tumores embrionarios o tumores neuroectodérmicos primitivos (PNET)
son tumores indiferenciados de células redondas, con núcleo
hipercromático y escaso citoplasma, alto cociente núcleo/citoplásmico y
diferentes patrones de diferenciación (neuronal, glial, ependimario…).
Representan un grupo heterogéneo de tumores del sistema nervioso
central (SNC) y del periférico (SNP), y tanto su origen como su
clasificación histopatológica no están claras. El meduloblastoma (MB)
es el PNET del SNC más común en la edad pediátrica, suponiendo el
20-25% de los tumores cerebrales. Los PNET supratentoriales son
histológicamente similares a los meduloblastomas, pero su pronóstico es
más agresivo que el de éstos. El tumor embrionario más frecuente del
SNP es el neuroblastoma (NB), que pertenece al grupo de los tumores neuroblásticos y presenta características histológicas
similares a los PNET centrales con diferenciación neuronal. Si bien los
MB y los PNET supratentoriales pertenecen al grado IV de la OMS, los tumores neuroblásticos pueden
presentar grados entre el I y el IV, aunque la clasificación del
International Neuroblastoma Pathology Committee se sigue más que la de la OMS en el caso de los tumores neuroblásticos,
con la idea de dar una clara orientación de pronóstico. Es una
clasificación basada en el grado de diferenciación neuroblástico y la
cantidad de estroma de Schwan. En esa clasificación se proponen cuatro
tipos de tumores neuroblásticos, ordenados a continuación en orden de
peor a mejor pronóstico: neuroblastoma, ganglioneuroblastoma nodular,
ganglioneuroblastoma entremezclado, y ganglioneuroma A nivel genético el MB se caracteriza por la presencia del isocromosoma 17q [i(17q)], así como por pérdidas en 17p, 10q y 1q, y ganancias en el cromosoma 7. Los NB se caracterizan por la amplificación de MYCN, las pérdidas de 1p y 11q, y las ganancias de 17q. Los PNET supratentoriales, por el contrario, carecen de definición genética, debido al escaso número de estudios realizados específicamente en este tipo de tumores. Generalmente, la bibliografía nos muestra estudios de los tumores embrionarios del sistema nervioso bajo el nombre genérico de PNET, pero casi la totalidad de los mismos se refieren específicamente a MB, y no a PNET supratentoriales. Por todo ello, el estudio presente pretende demostrar las diferencias que, a nivel genético pudieran existir entre los PNET supratentoriales y los MB, hasta ahora incluidos en un mismo grupo de clasificación genética. Además hemos estudiado mediante CGH (hibridación genómica comparada), un grupo de tumores neuroblásticos de varios tipos histológicos, a fin de determinar el perfil de pérdidas y ganancias de loci cromosómicos en los NB (tumores embrionarios del SNP), frente a los MB y PNET supratentoriales (tumores embrionarios del SNC).
|
|
|
|
Se estudiaron un total de 37 MB, 15 PNET supratentoriales y 26 tumores neuroblásticos (16 neuroblastomas, 7 ganglioneuroblastomas y 3 ganglioneuromas), además de 7 líneas celulares de MB y PNET, y DNA de sangre de 30 donantes sanos, según se indica en la Figura 1. El diseño experimental (Figura 2 a Figura 5) se hizo de tal manera que los tumores incluidos en parafina y los congelados se utilizaron para técnicas distintas. Así, los incluidos en parafina se sometieron a ensayos de PCR diferencial para el análisis de deleciones homocigóticas, y a ensayos de MSP (PCR específica de metilación) para el análisis de hipermetilación de promotores, mientras que los tumores congelados se sometieron a análisis de CGH (hibridación genómica comparada) para la determinación global de ganancias y pérdidas de material genético a nivel cromosómico. Las líneas celulares se emplearon sobre todo para el análisis de expresión, ya que no se disponía de RNA de los tumores. Los genes sometidos a estudio fueron genes localizados en el cromosoma 10q, en concreto PTEN (no confundir con PNET) y DMBT1; el locus CDKN2A (con dos genes: p14ARF y p16INK4A) del cromosoma 9p; y el gen RASSF1A, del cromosoma 3p. Todos estos brazos o loci cromosómicos participan en la génesis o progresión de diversos tumores cerebrales, como astrocitomas o MB. Las Figuras 6-8 incluyen esquemas de comprensión de las técnicas utilizadas.
|
|
|
|
Los resultados correspondientes a los experimentos de determinación de deleciones homocigóticas, hipermetilación de promotores y expresión a nivel de RNA se encuentran resumidos en las Figuras 9-11. CGH (Figura 12 a Figura 17): La alteración más frecuente en los PNET centrales fue la pérdida del cromosoma 17p, en 9 casos (45%), seguida de la ganancia de los cromosomas 1q, 7 y 17q, y de la pérdida del 16p en 7 casos (35%). La ganancia de 17q se dio en 8 casos de tumores neuroblásticos (35%). Otras alteraciones fueron ganancia del cromosoma 7, pérdida del 11q en 6 casos (26%), y ganancia a nivel del locus MYCN en 5 casos (22%). La ganancia de 1p, pérdida de 8q, ganancia de 9p, y pérdida de 17p mostraron asociación estadísticamente significativa con los PNET centrales (p<0.05). Al comparar los perfiles CGH de los PNET supratentoriales y los meduloblastomas, se detectó que en meduloblastomas la alteración más frecuente fue la pérdida de 17p (50%), asociada generalmente a ganancia de 17q (36%), lo que sugiere la formación de un isocromosoma 17q [i(17q)] que no ha sido detectado en PNET supratentoriales. La pérdida de 10q fue más frecuente en meduloblastomas (43%), sugiriendo que algunos genes localizados en este brazo cromosómico (PTEN, DMBT1) podrían ser de interés en la patogenia de los meduloblastomas, pero no de los PNET supratentoriales. Las diferencias en los perfiles genéticos entre meduloblastomas y PNET supratentoriales no fueron significativas.
|
|
|
|
Deleciones homocigóticas 1. Nuestros resultados, aunque preliminares, podrían mostrar que las deleciones homocigóticas de PTEN y DMBT1 contribuyen al desarrollo de una parte de PNET supratentoriales y MB de la siguiente manera: a. Los MB presentan más alteraciones genéticas en PTEN y DMBT1 que los PNET supratentoriales. b. Los PNET supratentoriales tienen alteraciones en DMBT1 pero no en PTEN c. Los MB presentan una frecuencia similar de alteraciones de PTEN y DMBT1. No obstante, no se han demostrado diferencias significativas, debido probablemente al pequeño número de PNET supratentoriales comparado con el de MB. 2. No se detectaron deleciones homocigóticas de p14ARF o p16INK4 en niguno de los MB y PNET supratentoriales estudiados. Hipermetilación de promotores 3. El estudio de hipermetilación de promotores podría ser de interés para subdividir los PNET centrales en cuanto a su estado de metilación: a. La hipermetilación de PTEN parece asociarse a los MB. b. La hipermetilación de p16INK4 es poco frecuente en MB y PNET supratentoriales. c. La inactivación epigenética de p14ARF podría constituir un evento genético de interés en la tumorigénesis de los PNET supratentoriales, ya que se detectó en alta frecuencia en PNET supratentoriales, siendo las diferencias significativas con respecto a la metilación encontrada en MB. d. La frecuencia de metilación del promotor de RASSF1A fue la más alta encontrada en nuestro estudio, tanto en PNET supratentoriales como en MB. Estudio de expresión 4. Los resultados del estudio de expresión en líneas celulares se correlacionan con los resultados encontrados en tumores. a. Ninguna de las líneas celulares de PNET analizadas expresaban RASSF1A. Estos resultados son concordandes con los del estudio de hipermetilación. b. Se confirma la alta frecuencia de pérdida de expresión de DMBT1 en líneas celulares de PNET, sugiriéndose un importante papel de este gen en el desarrollo de estos tumores. CGH 5. Hemos detectado diferencias en el perfil genético de ganancias y pérdidas de DNA entre MB y PNET supratentoriales. a. El isocromosoma 17q se detecta frecuentemente en MB, mientras que se observa raramente en PNET supratentoriales. b. Las ganancias de 1q son más frecuentes en PNET supratentoriales que en MB. c. En MB se demostraron otras alteraciones como ganancias del cromosoma 7 y regiones del 12 así como pérdidas a nivel de los cromosomas 10, 16q y 18q. Por el contrario, los PNET supratentoriales presentaron ganancias en los cromosomas 5, 9p y 18q, así como pédidas en 9p, 16p, 19p y en el cromosoma 22. d. Detectamos una nueva región de amplificación en dos casos de MB a nivel de 4q12, que requiere un estudio posterior más profundo. 6. Las ganancias de 1q y 9p, y las pérdidas de 8q y 17p se asocian significativamente a los PNET centrales (MB y PNET supratentoriales) y no a los PNET periféricos (tumores neuroblásticos estudiados). El presente trabajo, en conjunto, confirma la hipótesis de que los tumores embrionarios del sistema nervioso constituyen diferentes entidades genéticas con diferentes alteraciones moleculares, independientemente de la apariencia histológica similar que presentan.
|
|
|
|
La Dra. María del Mar Inda agradece al Ministerio de Educación Cultura
y Deporte la concesión de una beca predoctoral para la realización de
este trabajo de investigación, cuya financiación experimental se ha
llevado a cabo gracias a fondos del Ministerio de Ciencia y Tecnología,
Fondo de Investigación Sanitaria, Departamento de Salud del Gobierno de
Navarra, Fundación Científica de la Asociación Contra el Cáncer, y
Fundación Universidad de Navarra.
|
|
|
|
1. Kleihues P, Cavenee WK. Pathology and genetics of tumours of the nervous system. Lyon: IARC Press, 2000. 2. McComb RD, Burger PC. Pathologic analysis of primary brain tumors. Neurol Clin 1985; 3; 711-728. 3. Paulino AC, Melian E. Medulloblastoma and supratentorial primitive neuroectodermal tumors: an institutional experience. Cancer 1999; 86; 142-148. 4. Burnett ME, White EC, Sih S, von Haken MS, Cogen PH. Chromosome arm 17p deletion analysis reveals molecular genetic heterogeneity in supratentorial and infratentorial primitive neuroectodermal tumors of the central nervous system. Cancer Genet Cytogenet 1997; 97; 25-31. 5. Fruhwald MC, O'Dorisio MS, Dai Z et al. Aberrant hypermethylation of the major breakpoint cluster region in 17p11.2 in medulloblastomas but not supratentorial PNETs. Genes Chromosomes Cancer 2001; 30; 38-47. 6. Russo C, Pellarin M, Tingby O et al. Comparative genomic hybridization in patients with supratentorial and infratentorial primitive neuroectodermal tumors. Cancer 1999; 86; 331-339. 7. Biegel JA, Rorke LB, Packer RJ et al. Isochromosome 17q in primitive neuroectodermal tumors of the central nervous system. Genes Chromosomes Cancer 1989; 1; 139-147. 8. Pruchon E, Chauveinc L, Sabatier L et al. A cytogenetic study of 19 recurrent gliomas. Cancer Genet Cytogenet 1994; 76; 85-92. 9. Biegel JA. Cytogenetics and molecular genetics of childhood brain tumors. Neuro-oncol 1999; 1; 139-151. 10. Bigner SH, Mark J, Friedman HS, Biegel JA, Bigner DD. Structural chromosomal abnormalities in human medulloblastoma. Cancer Genet Cytogenet 1988; 30; 91-101. 11. Badiali M, Pession A, Basso G et al. N-myc and c-myc oncogenes amplification in medulloblastomas. Evidence of particularly aggressive behavior of a tumor with c-myc amplification. Tumori 1991; 77; 118-121. 12. Batra SK, Rasheed BK, Bigner SH, Bigner DD. Oncogenes and anti-oncogenes in human central nervous system tumors. Lab Invest 1994; 71; 621-637. 13. Bigner SH, Humphrey PA, Wong AJ et al. Characterization of the epidermal growth factor receptor in human glioma cell lines and xenografts. Cancer Res 1990; 50; 8017-8022. 14. Troost D, Jansen GH, Dingemans KP. Cerebral medulloepithelioma--electron microscopy and immunohistochemistry. Acta Neuropathol 1990; 80; 103-107. 15. Adesina AM, Nalbantoglu J, Cavenee WK. p53 gene mutation and mdm2 gene amplification are uncommon in medulloblastoma. Cancer Res 1994; 54; 5649-5651. 16. Ohgaki H, Eibl RH, Schwab M et al. Mutations of the p53 tumor suppressor gene in neoplasms of the human nervous system. Mol Carcinog 1993; 8; 74-80. 17. Mollenhauer J, Wiemann S, Scheurlen W et al. DMBT1, a new member of the SRCR superfamily, on chromosome 10q25.3-26.1 is deleted in malignant brain tumours. Nat Genet 1997; 17; 32-39. 18. Inda MM, Mercapide J, Munoz J et al. PTEN and DMBT1 homozygous deletion and expression in medulloblastomas and supratentorial primitive neuroectodermal tumors. Oncol Rep 2004; 12; 1341-1347. 19. Stott FJ, Bates S, James MC et al. The alternative product from the human CDKN2A locus, p14(ARF), participates in a regulatory feedback loop with p53 and MDM2. Embo J 1998; 17; 5001-5014. 20. Fan X, Inda MM, Tunon T, Castresana JS. Improvement of the methylation specific PCR technical conditions for the detection of p16 promoter hypermethylation in small amounts of tumor DNA. Oncol Rep 2002; 9; 181-183. 21. Herman JG, Graff JR, Myohanen S, Nelkin BD, Baylin SB. Methylation-specific PCR: a novel PCR assay for methylation status of CpG islands. Proc Natl Acad Sci U S A 1996; 93; 9821-9826. 22. Barker FG, Chen P, Furman F, Aldape KD, Edwards MS, Israel MA. p16 deletion and mutation analysis in human brain tumors. J Neurooncol 1997; 31; 17-23. 23. Sato K, Schauble B, Kleihues P, Ohgaki H. Infrequent alterations of the p15, p16, CDK4 and cyclin D1 genes in non-astrocytic human brain tumors. Int J Cancer 1996; 66; 305-308. 24. Jen J, Harper JW, Bigner SH et al. Deletion of p16 and p15 genes in brain tumors. Cancer Res 1994; 54; 6353-6358. 25. Raffel C, Ueki K, Harsh GRt, Louis DN. The multiple tumor suppressor 1/cyclin-dependent kinase inhibitor 2 gene in human central nervous system primitive neuroectodermal tumor. Neurosurgery 1995; 36; 971-974. 26. Kamiryo T, Tada K, Shiraishi S et al. Analysis of homozygous deletion of the p16 gene and correlation with survival in patients with glioblastoma multiforme. J Neurosurg 2002; 96; 815-822. 27. Nakamura M, Watanabe T, Klangby U et al. p14ARF deletion and methylation in genetic pathways to glioblastomas. Brain Pathol 2001; 11; 159-168. 28. Waha A, Koch A, Meyer-Puttlitz B et al. Epigenetic silencing of the HIC-1 gene in human medulloblastomas. J Neuropathol Exp Neurol 2003; 62; 1192-1201. 29. Ebinger M, Senf L, Wachowski O, Scheurlen W. Promoter Methylation Pattern of Caspase-8, P16INK4A, MGMT, TIMP-3, and E-Cadherin in Medulloblastoma. Pathol Oncol Res 2004; 10; 17-21. 30. Fruhwald MC, O'Dorisio MS, Dai Z et al. Aberrant promoter methylation of previously unidentified target genes is a common abnormality in medulloblastomas--implications for tumor biology and potential clinical utility. Oncogene 2001; 20; 5033-5042. 31. Harada K, Toyooka S, Maitra A et al. Aberrant promoter methylation and silencing of the RASSF1A gene in pediatric tumors and cell lines. Oncogene 2002; 21; 4345-4349. 32. Lindsey JC, Lusher ME, Anderton JA et al. Identification of tumour-specific epigenetic events in medulloblastoma development by hypermethylation profiling. Carcinogenesis 2004; 25; 661-668. 33. Gonzalez-Gomez P, Bello MJ, Lomas J et al. Epigenetic changes in pilocytic astrocytomas and medulloblastomas. Int J Mol Med 2003; 11; 655-660. 34. María del Mar Inda, Jorge Muñoz, Philippe Coullin, et al. High promoter hypermethylation frequency of p14/ARF in supratentorial PNET but not in medulloblastoma. Histopathology (in press) 35. María del Mar Inda, Christine Perot, Marine Guillaud-Bataille, et al. Genetic heterogeneity in supratentorial and infratentorial primitive neuroectodermal tumors of the central nervous system Histopathology (in press).
|
|
|
|
- Silvia Garcia Martinez (25/10/2005 22:24:47)
- Zurama Eloisa Castro Castro (27/10/2005 2:57:19)
|
|
|
|
|
Web mantenido y actualizado por el Servicio de informática uclm. Modificado: 16/06/2015 14:10:50
.jpg) fiogf49gjkf0dFigura 1: Material tumoral, líneas celulares y controles sometidos a estudio.">
fiogf49gjkf0dFigura 1: Material tumoral, líneas celulares y controles sometidos a estudio.">
.jpg) fiogf49gjkf0dFigura 2: Diseño experimental para el análisis de deleciones homocigóticas mediante PCR diferencial.">
fiogf49gjkf0dFigura 2: Diseño experimental para el análisis de deleciones homocigóticas mediante PCR diferencial.">
.jpg) fiogf49gjkf0dFigura 3: Diseño experimental para el análisis de hipermetilación de promotores mediante PCR específica de metilación (MSP).">
fiogf49gjkf0dFigura 3: Diseño experimental para el análisis de hipermetilación de promotores mediante PCR específica de metilación (MSP).">
.jpg) fiogf49gjkf0dFigura 4: Diseño experimental para el análisis de expresión génica mediante RT-PCR.">
fiogf49gjkf0dFigura 4: Diseño experimental para el análisis de expresión génica mediante RT-PCR.">
.jpg) fiogf49gjkf0dFigura 5: Diseño experimental para el análisis de pérdidas y ganancias de DNA a nivel cromosómico mediante CGH (hibridación genómica comparada).">
fiogf49gjkf0dFigura 5: Diseño experimental para el análisis de pérdidas y ganancias de DNA a nivel cromosómico mediante CGH (hibridación genómica comparada).">
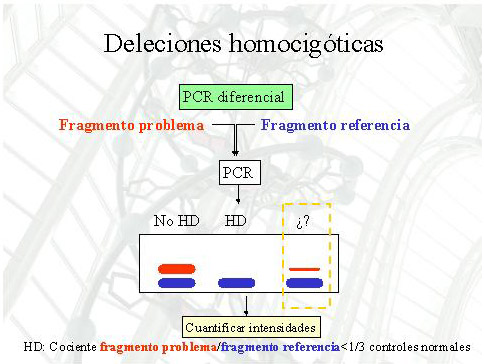 fiogf49gjkf0dFigura 6: Esquema técnico para el análisis de deleciones homocigóticas mediante PCR diferencial.">
fiogf49gjkf0dFigura 6: Esquema técnico para el análisis de deleciones homocigóticas mediante PCR diferencial.">
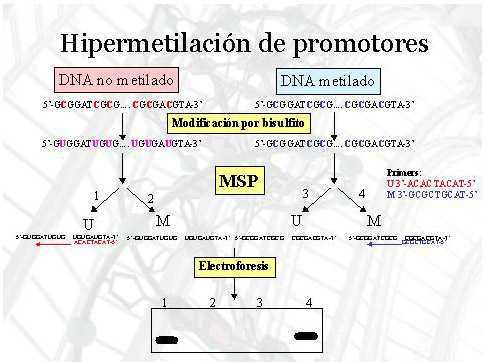 fiogf49gjkf0dFigura 7: Esquema técnico para el análisis de hipermetilación de promotores mediante MSP (PCR específica de metilación).">
fiogf49gjkf0dFigura 7: Esquema técnico para el análisis de hipermetilación de promotores mediante MSP (PCR específica de metilación).">
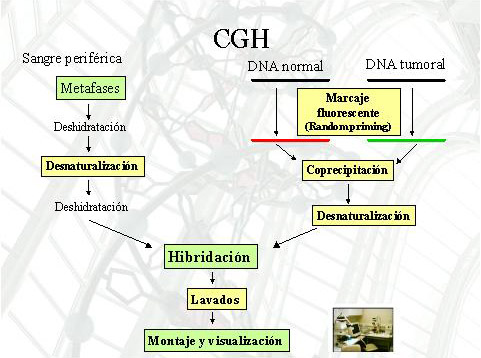 fiogf49gjkf0dFigura 8: Esquema técnico para el análisis de pérdidas y ganancias de DNA a nivel cromosómico mediante CGH (hibridación genómica comparada).">
fiogf49gjkf0dFigura 8: Esquema técnico para el análisis de pérdidas y ganancias de DNA a nivel cromosómico mediante CGH (hibridación genómica comparada).">
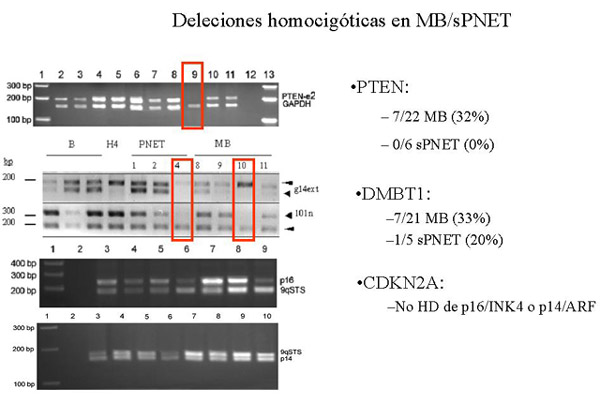 fiogf49gjkf0dFigura 9: Resultados de deleciones homocigóticas en los genes PTEN y DMBT1 y en el locus CDKN2A (genes p14ARF y p16INK4)">
fiogf49gjkf0dFigura 9: Resultados de deleciones homocigóticas en los genes PTEN y DMBT1 y en el locus CDKN2A (genes p14ARF y p16INK4)">
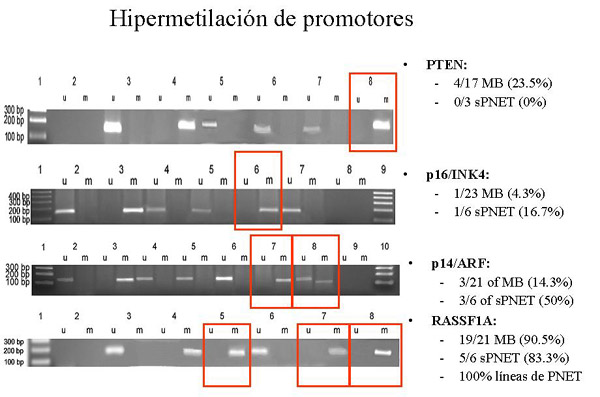 fiogf49gjkf0dFigura 10: Resultados de hipermetilación de promotores en los genes PTEN, p14ARF, p16INK4 y RASSF1A.">
fiogf49gjkf0dFigura 10: Resultados de hipermetilación de promotores en los genes PTEN, p14ARF, p16INK4 y RASSF1A.">
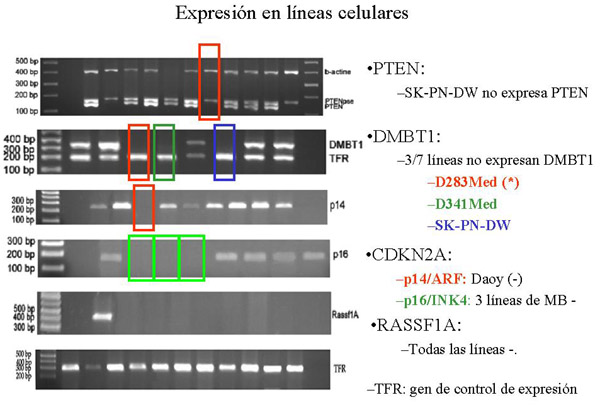 fiogf49gjkf0dFigura 11: Resultados de expresión a nivel de RNA de los genes PTEN, DMBT1, p14ARF, p16INK4 y RASSF1A, en líneas celulares mediante RT-PCR">
fiogf49gjkf0dFigura 11: Resultados de expresión a nivel de RNA de los genes PTEN, DMBT1, p14ARF, p16INK4 y RASSF1A, en líneas celulares mediante RT-PCR">
.jpg) fiogf49gjkf0dFigura 12: Ejemplo de metafase para CGH. Las flechas verdes indican ganancias; las rojas, pérdidas; y las amarillas, normalidad en el número de copias de DNA.">
fiogf49gjkf0dFigura 12: Ejemplo de metafase para CGH. Las flechas verdes indican ganancias; las rojas, pérdidas; y las amarillas, normalidad en el número de copias de DNA.">
.jpg) fiogf49gjkf0dFigura 13: Ejemplo de un cariotipo para CGH. Corresponde a un caso de DNA tumoral. Se aprecian claramente los colores verdes (ganancias) y rojos (pérdidas).">
fiogf49gjkf0dFigura 13: Ejemplo de un cariotipo para CGH. Corresponde a un caso de DNA tumoral. Se aprecian claramente los colores verdes (ganancias) y rojos (pérdidas).">
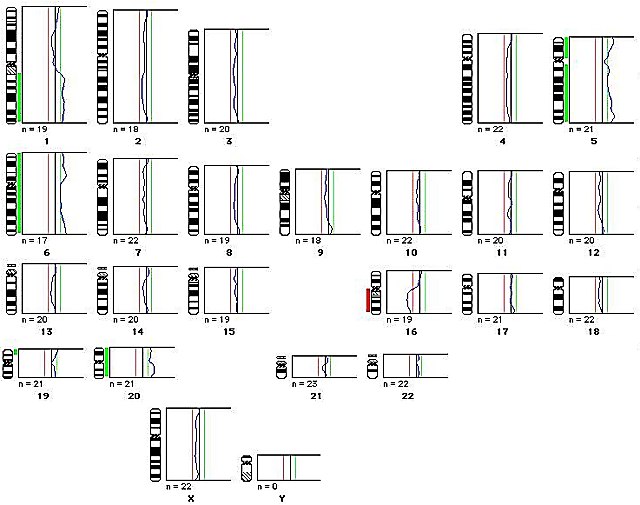 fiogf49gjkf0dFigura 14:
fiogf49gjkf0dFigura 14: 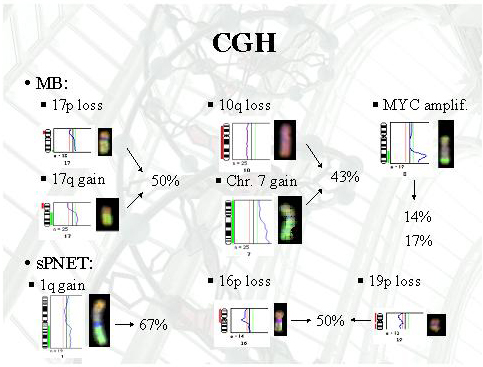 fiogf49gjkf0dFigura 15: Resultados del análisis mediante CGH en MB y en PNET supratentoriales, mostrándose las alteraciones más frecuentes en cada uno de ellos.">
fiogf49gjkf0dFigura 15: Resultados del análisis mediante CGH en MB y en PNET supratentoriales, mostrándose las alteraciones más frecuentes en cada uno de ellos.">
.jpg) fiogf49gjkf0dFigura 16: Comparación de lesiones detectables mediante CGH y frecuencia de las mismas, en MB y PNET supratentoriales">
fiogf49gjkf0dFigura 16: Comparación de lesiones detectables mediante CGH y frecuencia de las mismas, en MB y PNET supratentoriales">
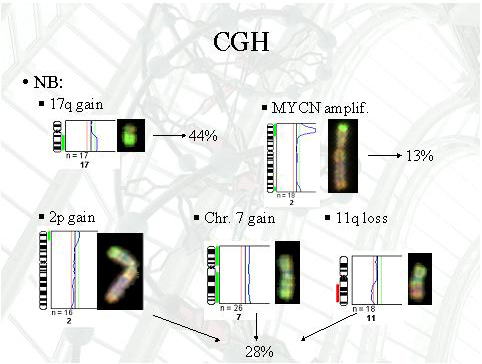 fiogf49gjkf0dFigura 17: Resultados del análisis de CGH en tumores neuroblásticos.">
fiogf49gjkf0dFigura 17: Resultados del análisis de CGH en tumores neuroblásticos.">