

|
Joaquín Jaén Martínez*, Marylène Lejeune*, Lluis E. Pons Ferré*, Maria Teresa Salvadó Usach*, Marcial García Rojo**, Carlos Lopez Pablo*, Jordi Baucells Lluís***, Xavier Cugat Mata***, Ramón Bosch Príncep*, Tomás Álvaro Naranjo* |
|
|
Introducción: La evaluación actual de los resultados de técnicas inmunohistoquímicas por parte del patólogo conlleva inherente una serie de limitaciones respecto al tiempo invertido, a la subjetividad de la interpretación y reproducibilidad de resultados dado el elevado número de factores que pueden interferir en el resultado final de la técnica. El empleo de software comercial para el análisis digital de imágenes basado en procesos de segmentación se ha destacado como un instrumento capaz de minimizar tales problemas. Sin embargo, existen limitaciones específicas resultante de la falta de sistemas de referencia bien definidos (un área) para realizar la cuantificación, la falta de consenso respecto a la unidad susceptible de contabilización (células teñidas o áreas de tinción) o la propia variabilidad respecto a la localización celular del marcador a identificar (nuclear, citoplasmática, membrana). Todo lo cual justifica el desarrollo y mejora de los sistemas de análisis de imagen digital existentes en la actualidad.
Material y Métodos: Estudio retrospectivo de proteínas representativas de diferentes patrones de localización celular en linfomas de Hodgkin mediante la construcción de bloques multitejido. Realización de técnicas inmunohistoquímicas para la detección de antígenos nucleares, citoplasmáticos y de membrana (CD4, CD8, CD56, CD68, HLA-DR, TIA-1, p53, Ki67). Captura de un total de 196 imágenes representativas mediante microscopio óptico (40x) con cámara digital incorporada (Coolsnap Photometrics). Evaluación del numero de células teñidas en cada imagen mediante doble cuantificación manual por parte de tres observadores diferentes y cuantificación semiautomática mediante el software de análisis de imagen Image-Pro Plus® 5.0, complementado por macros específicas desarrolladas en nuestro laboratorio capaces de cuantificar células marcadas en base a algoritmos que permiten la automatización respecto al umbral de color específico y rasgos morfológicos. Análisis estadístico utilizando SPSS Statistical Software 11.1®. Resultados: El análisis de los resultados correspondiente a la cuantificación manual pone de manifiesto una variabilidad intraobservador, especialmente significativa cuando la lectura se efectúa sobre técnicas que identifican proteínas de localización citoplasmática y de membrana. Sin embargo, las comparaciones interobservadores señalan una clara disminución de estas discordancias aunque las diferencias entre las mediciones siguen siendo notables. La reproducibilidad de la cuantificación de las células teñidas en una misma imagen utilizando las macros del software es plena. La comparación entre estos resultados y los obtenidos manualmente, muestra que el análisis semiautomático proporciona un recuento en valores absolutos inferior al método manual. No obstante, la discordancia entre los resultados obtenidos por el procedimiento manual y semiautomático aumenta de forma directamente proporcional al número de células positivas encontradas por campo. Discusión: El empleo de sistemas de análisis digital de imágenes aplicado a la evaluación de técnicas inmunohistoquímicas presenta mayor precisión, rapidez y reproducibilidad frente a la cuantificación manual actual. No obstante, la aplicación de macros específicas según la localización de la proteína a evaluar no está exenta de limitaciones técnicas, en nuestro caso en la evaluación de proteínas de localización de membrana en imágenes de alta densidad celular. Se requiere, por tanto, el desarrollo y mejora de los algoritmos necesarios para la identificación precisa de los marcadores a estudio. Trabajo financiado por el Fondo de Investigación Sanitaria (FIS 04/1440 y FIS 04/1467). Instituto de Salud Carlos III. Ministerio de Sanidad y Consumo.
|
||
|
|
La evaluación actual de los resultados de técnicas inmunohistoquímicas por parte del patólogo conlleva inherente una serie de limitaciones respecto al tiempo invertido, a la subjetividad de la interpretación y reproducibilidad de resultados dado el elevado número de factores que pueden interferir en el resultado final de la técnica (1, 2, 3). Dichos factores pueden depender tanto de la propia técnica de inmunotinción, como del tipo de anticuerpo empleado y su dilución, del sistema de detección o del rescate antigénico; factores todos ellos considerados modificables y susceptibles de estandarización intra e interlaboratorio. Un segundo grupo de variables estaría integrado por factores relacionados con el proceso de fijación tisular (tipo de fijador, tiempo de fijación, método), procesamiento tisular y la preservación y expresión antigénica intrínseca de cada tejido. Estos últimos factores, de notable impacto en el resultado final, están sujetos a frecuente variación, resultando más problemática su estandarización (4).
El empleo de software comercial para el análisis digital de imágenes basado en procesos de segmentación se ha destacado como un instrumento capaz de minimizar tales problemas (5, 6). Sin embargo, existen limitaciones específicas resultante de la falta de sistemas de referencia bien definidos para realizar la cuantificación, la falta de consenso respecto a la unidad susceptible de contabilización (células teñidas o áreas de tinción) o la propia variabilidad respecto a la localización celular del marcador a identificar (nuclear, citoplasmática, membrana) (7, 8). Todo lo cual justifica el desarrollo y mejora de los sistemas de análisis de imagen digital existentes en la actualidad. En el presente estudio, utilizando el software Imagen-Pro Plus® 5.0 (Media Cybernetics, U.S.A.), hemos desarrollado diversas macros basadas en el reconocimiento de rasgos de imagen para identificar las células positivas teñidas con el cromógeno DAB. Estos algoritmos permiten aislar, de la forma más precisa posible, las áreas positivas a partir de la automatización del umbral especifico de color de tinción (tonos marrones y azules, luminosidad y saturación) y de diversas características morfológicas. El propósito principal es la obtención de macros específicas según la localización del anticuerpo (patrón de tinción) que evite la problemática resultante del incremento progresivo del número de marcadores que precisan una cuantificación específica y la comparación de resultados respecto a la evaluación manual realizada actualmente.
|
|
|
|
Estudio retrospectivo realizado mediante la construcción de bloques multitejido (TMA), según técnica descrita previamente (9), a partir de bloques de parafina de 196 casos de Linfoma de Hodgkin (LH) seleccionados e identificados de manera aleatoria por el Grupo Español de Estudio del Linfoma de Hodgkin entre 1994 y 1998.
Figura 2. Búsqueda de las áreas representativas con los objetivos de 10-20x y captura de la imagen elegida a 40x.">
Selección de anticuerpos representativos de patrón de localización de membrana: CD4, CD8, CD56 (Novocastra; Newcastle, UK), HLA-Dr (DakoCytomation; Carpintería, USA); de patrón de localización citoplasmática: TIA-1 (Master Diagnóstica;Granada, España), CD68 (DakoCytomation; Carpintería, USA); y de patrón de localización nuclear: Ki67, P53 (DakoCytomation; Carpintería, USA). Proceso de inmunotinción automatizado mediante robot Horizon TechMate, siguiendo instrucciones de los fabricantes, como se ha descrito previamente (10). Selección de áreas representativas, previa valoración de los resultados de la técnica inmunohistoquímica (controles), mediante la identificación de las zonas de mayor densidad de células positivas con presencia de células tumorales acompañadas de fondo inflamatorio apropiado, evitando focos desvitalizados o con artefacto. Captura de imágenes mediante microscopio óptico Zeiss Axioskop 2 plus (Carl Zeiss; Gvttingen, Germany) con cámara digital Coolsnap acoplada (Photometrics; Tucson, USA) y conectada a ordenador Pentium III con sistema operativo Windows 2000, complementado con el software de análisis de imagen Image-pro Plus 5.0® (Figura 1). Las imágenes fueron obtenidas con el objetivo 40x, con una resolución 1392x1040 píxeles en RGB 24 y almacenadas en formato TIFF. En total fueron obtenidas 196 imágenes de las cuales 100 correspondían al patrón de tinción de membrana [CD4 (n=30), CD8 (n=32), CD56 (n=19) y HLA-Dr (n=19)], 66 correspondían al patrón de tinción del citoplasma [CD68 (n=28) y TIA-1 (n=38)] y finalmente, 30 que correspondían al patrón de tinción nuclear [KI-67 (n=15) y p53 (n=15)]. La superficie real de cada campo analizado fue de 0.04 mm2 (Figura 2). La cuantificación manual fue realizada por parte de tres observadores diferentes (dos patólogos y un biólogo), con un mes de intervalo entre las dos lecturas efectuadas. Las células consideradas positivas en cada una de las imágenes fueron cuantificadas directamente en la pantalla mediante la función “Manual Tag” del software Image-Pro Plus® (Figura 3). La cuantificación semiautomática fue realizada mediante el citado software, complementado por macros específicas desarrolladas en nuestro laboratorio. Básicamente, estas macros consisten en una serie de pasos sucesivos que permiten la evaluación automática de los diversos patrones de tinción (Figura 4). En primer lugar, optimizamos las características morfométricas de la imagen para eliminar la contribución del ruido de fondo, excluir los detritus celulares y resaltar el color positivo. Esta mejora de la imagen se realiza modificando el ajuste de brillo, contraste y gamma en los canales RGB (rojo, verde y azul). El segundo lugar determinamos los umbrales de color, tamaño y forma de las células teñidas usando el comando de "Count/size" del programa para cada patrón de tinción. En este proceso, la intensidad del color de los objetos positivos (marrón) fue preestablecida manualmente para cada patrón (píxel por píxel) basándose en el histograma HSI (tonalidad-saturación-intensidad). La calibración preestablecida de las imágenes permiten definir el área media de las células positivas y así eliminar los eventuales clusters (agrupaciones de células). La opción final del comando "Count/size" nos permite obtener el numero de células positivas en cada imagen, valor que es automáticamente transferido a un hoja de cálculo (Excel). Los algoritmos correspondientes a cada uno de estos comandos fueron integrados en sus macros respectivas. El análisis estadístico se ha realizado mediante el software estadístico SPSS 11.1®. En primer lugar, se ha valorado la reproducibilidad intraobservador (grado de coincidencia que mantiene un observador consigo mismo al valorar la misma muestra) y la reproducibilidad interobservadores (grado de coincidencia entre dos o más observadores que valoran la misma muestra). El análisis de concordancia fue realizado mediante el coeficiente de correlación y el coeficiente de correlación intraclase (ICC), con sus correspondientes representaciones gráficas. En segundo lugar se ha valorado la reproducibilidad entre el método manual y el método automático. La validez del método automático fue determinado igualmente vía coeficiente de correlación intraclase y representación gráfica según el método de Bland-Altman. Figura 1. Bloques multitejido (TMA), inmunohistoquímica, estación de análisis de imágenes."> Figura 1 - Figura 1. Bloques multitejido (TMA), inmunohistoquímica, estación de análisis de imágenes.
Figura 2 - Figura 2. Búsqueda de las áreas representativas con los objetivos de 10-20x y captura de la imagen elegida a 40x.
|
|
|
|
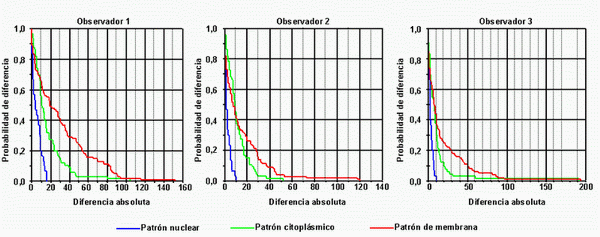
La evaluación de los resultados correspondiente al recuento manual pone de manifiesto la existencia de variabilidad tanto intra como inter-observador. La variabilidad intra-observador, resultante de las diferencias existentes entre la primera y segunda lectura efectuada por cada uno de los observadores, se detecta en los tres patrones de inmunotinción preestablecidos, pero es especialmente significativa en la lecturas correspondientes a la localización citoplasmática y de membrana (Figura 5). Por su parte, la variabilidad ínter-observador (consecuencia de la comparación entre las medias de las dos lecturas de cada observador), sólo muestra variabilidad estadísticamente significativa en las lecturas de marcadores de localización citoplasmática y de membrana pero no respecto a las técnicas de localización nuclear (Figura 6).
Figura 6. Curva de Kaplan-Meier (análisis inter-observador): Probabilidad de tener una diferencia en valor absoluto entre mediciones realizadas por observadores diferentes para tinciones con patrón nuclear, citoplásmicos y de membrana. En el 80% de los casos hay una diferencia máxima de 10 células para el patrón nuclear, de 10 células para el patrón citoplásmico y de 35 células para el patrón de membrana.">
La representación gráfica de Bland-Altman de las diferencias entre las dos mediciones opuestas a su promedio, nos demuestra que estas diferencias no son homogéneas y que la discordancia es más alta cuando las imágenes presentan una gran cantidad de células positivas por campo (>100 células/campo) (Figura 7). La cuantificación semi-automática se realiza de forma rápida. El tiempo necesario para la realización del recuento (imagen en la pantalla, aplicación de la macro y transferencia de los datos a Excel) asciende a 5-6 segundos por imagen, siendo plena la reproducibilidad de los resultados. Cuando comparamos los recuentos obtenidos mediante el software y los obtenidos de forma manual se evidencia que el análisis semiautomático proporciona unos resultados en valores absolutos inferiores al método manual y discordancia en la cuantificación de proteínas con patrón de tinción de membrana, sobre todo en imágenes con alta densidad de células positivas (Figura 8 y Figura 9). Figura 5. Curvas de Kaplan-Meier (análisis intra-observador): Probabilidad de tener una diferencia en valor absoluto entre las dos mediciones de cada uno de los tres observadores para tinciones con patrones nucleares, citoplásmicos y de membrana. En el 80% de los casos hay una diferencia máxima de 10 células por el patrón nuclear, de 20 células por el patrón citoplásmico y de 50 células por el patrón de membrana."> Figura 5 - Figura 5. Curvas de Kaplan-Meier (análisis intra-observador): Probabilidad de tener una diferencia en valor absoluto entre las dos mediciones de cada uno de los tres observadores para tinciones con patrones nucleares, citoplásmicos y de membrana. En el 80% de los casos hay una diferencia máxima de 10 células por el patrón nuclear, de 20 células por el patrón citoplásmico y de 50 células por el patrón de membrana.
Figura 6 - Figura 6. Curva de Kaplan-Meier (análisis inter-observador): Probabilidad de tener una diferencia en valor absoluto entre mediciones realizadas por observadores diferentes para tinciones con patrón nuclear, citoplásmicos y de membrana. En el 80% de los casos hay una diferencia máxima de 10 células para el patrón nuclear, de 10 células para el patrón citoplásmico y de 35 células para el patrón de membrana.
|
|
|
|
El empleo de técnicas de inmunohistoquímica para la detección de antígenos celulares específicos se ha convertido en una herramienta indispensable en el diagnóstico anatomopatológico actual, participando en la identificación de microorganismos, clasificación de tumores, demostración de micrometástasis, provisión de información pronóstica e identificación de dianas terapéuticas (11). Especial repercusión en la práctica clínica diaria ha adquirido la identificación inmunohistoquímica de marcadores pronósticos tumorales (12) y de respuesta específica a las emergentes estrategias terapéuticas, precisando métodos de cuantificación de la inmunotinción que varían desde el recuento simple del número de células teñidas hasta la gradación de la intensidad acorde con escalas arbitrarias. En estos casos, la variabilidad inter e intraobservador es alta dado el elevado número de factores que pueden interferir en el resultado de la técnica (1). Dichos factores pueden depender tanto de la propia técnica de inmunotinción, como de factores relacionados con el proceso de fijación, procesamiento y expresión antigénica intrínseca de cada tejido, todos ellos sujetos a variación y susceptibles de diferente grado de estandarización intra e inter-laboratorio.
El impacto específico sobre el resultado final atribuido exclusivamente al proceso de interpretación por parte del observador ha sido previamente estudiado (2). La ausencia de estándares bien definidos respecto a qué debe ser considerado como un resultado positivo es una de las principales causas de la falta de reproducibilidad. En el presente estudio hemos establecido que, bajo condiciones de máxima estandarización y consenso respecto a criterios de positividad, la localización celular del anticuerpo a estudio y la densidad de células positivas influyen de forma significativa en la interpretación final de la técnica: la cuantificación de anticuerpos de localización citoplasmática y de membrana en áreas de elevada densidad de células positivas (>100 células positivas/0.04mm2) muestra las mayores diferencias. Un novedoso abordaje que, junto a la estandarización de técnicas, puede minimizar las limitaciones inherentes a las múltiples fases implicadas en el análisis cuantitativo de las técnicas inmunohistoquímicas consiste en el empleo de sistemas de análisis digital de imagen. En la actualidad existe en el mercado software diverso (ACIS, Image-Pro Plus, VIDS II, Qwin 500IW, ...) que permite el análisis de los resultados inmunohistoquímicos utilizando sistemas de procesamiento de imágenes basados en la segmentación de las mismas. En base a este proceso, diferentes trabajos recientes constatan una mayor precisión, sensibilidad y reproducibilidad de los sistemas de cuantificación semiautomática frente al recuento manual en diferentes tipos de tejidos (5, 6); aunque no exentos de áreas de conflicto pendientes de resolución como la falta de sistemas de referencia bien definidos (un área) para realizar la cuantificación, la falta de consenso respecto a la unidad susceptible de contabilización (células teñidas o áreas de tinción) o la propia variabilidad respecto a la localización celular del marcador a identificar (nuclear, citoplasmática, membrana). Todos ellos requieren unos tratamientos especiales con lo que el número de marcadores que precisan una cuantificación específica aumenta progresivamente (13). Constatada la influencia del patrón de tinción y conocida la aportación de los sistemas de análisis de imagen, desarrollamos los protocolos de aplicación de los algoritmos más adecuados (macros) para la evaluación cuantitativa según la localización del anticuerpo. La optimización de las características morfométricas de la imagen junto a la determinación los umbrales óptimos de color, tamaño y forma de las células teñidas para cada patrón de tinción permite una cuantificación rápida, precisa y reproducible. La cuantificación de anticuerpos con patrón de membrana en imágenes con alta densidad de células positivas y la inclusión de nuevos marcadores continua siendo objeto de estudio con la adaptación o desarrollo de los algoritmos matemáticos que nos permitan corregir los problemas detectados.
|
|
|
|
1 - Rhodes A, Jasani B, Anderson E, Dodson AR, Balaton AJ. Evaluation of HER-2/neu immunohistochemical assay sensitivity and scoring on formalin-fixed and paraffin-processed cell lines and breast tumors: a comparative study involving results from laboratories in 21 countries. Am J Clin Pathol 2002;118(3):408-417.
2 - Seidal T, Balaton AJ, Battifora H. Interpretation and quantification of immunostains. Am J Surg Pathol. 2001;25:1204-1207. 3 - Taylor CR. The total test approach to standardization of immunohistochemistry. Arch Pathol Lab Med 2000; 124:945-951. 4 - Leong FJ, Leong AS. Digital imaging in pathology: theoretical and practical considerations, and applications. Pathology. 2004;36:234-241. 5 - Hilbe W, Gachter A, Duba HC, Dirnhofer S, Eisterer W, Schmid T, et al. Comparison of automated cellular imaging system and manual microscopy for immunohistochemically stained cryostat sections of lung cancer specimens applying p53, ki-67 and p120. Oncol Rep 2003;10(1):15-20. 6 - Barreiro B, Badosa G, Quintana S, Esteban L, Heredia JL. Comparison between automatic and manual analysis in the diagnosis of obstructive sleep apnea-hypopnea syndrome. Arch Bronconeumol 2003;39(12):544-548. 7 - Camp RL, Chung GG, Rimm DL. Automated subcellular localization and quantification of protein expression in tissue microarrays. Nat Med 2002;8(11):1323-1327. 8 - Ross JS, Fletcher JA, Bloom KJ, Linette GP, Stec J, Symmans WF, et al. Targeted therapy in breast cancer: the HER-2/neu gene and protein. Mol Cell Proteomics 2004;3(4):379-398. 9 - Natkunam Y, Warnke RA, Montgomery K, Falini B, van De Rijn M. Analysis of MUM1/IRF4 protein expression using tissue microarrays and immunohistochemistry. Mod Pathol. 2001;14(7):686-694. 10 - Álvaro-Naranjo T, Lejeune M, Salvadó-Usach MT, Bosch-Príncep R, Reverter-Branchat G, Jaén-Martínez J, Pons-Ferré LE. Tumor-infiltrating cells as a prognostic factor in Hodgkin's lymphoma. A quantitative tissue microarray study in a large retrospective cohort of 267 patients. Leukemia & Lymphoma in press 2005. 11 - Chan JK. Advances in immunohistochemistry: impact on surgical pathology practice. Semin Diagn Pathol 2000;17(3):170-177. 12 - Oreskovic S, Babic D, Kalafatic D, Barisic D, Beketic-Oreskovic L. A significance of immunohistochemical determination of steroid receptors, cell proliferation factor Ki-67 and protein p53 in endometrial carcinoma. Gynecol Oncol 2004;93(1):34-40. 13 - Gil J, Wu HS. Applications of image analysis to anatomic pathology: realities and promises. Cancer Invest 2003;21(6):950-959.
|
|
|
|
- Julia Cecilia Cruz Mojarrieta (02/10/2005 15:08:34)
- Liliette Caraballoso García (09/10/2005 19:30:27)
- Riánsares Arriazu Navarro (09/10/2005 20:32:31)
- Joaquín Jaén Martínez (10/10/2005 16:28:24)
- Cesáreo Corbacho Cuevas (10/10/2005 18:15:11)
- ELSIE BEATRIZ PICOTT RANGEL (14/10/2005 8:24:22)
- MIGUEL ANGEL LIMERES GONZÁLEZ (23/10/2005 18:23:09)
- Nancy Ríos Hidalgo (26/10/2005 5:21:42)
- Joaquín Jaén Martínez (03/11/2005 13:23:38)
- OSCAR LOPEZ SANDOVAL (03/11/2005 14:27:37)
|
|
|
|
|
Web mantenido y actualizado por el Servicio de informática uclm. Modificado: 16/06/2015 14:10:50
 fiogf49gjkf0d
fiogf49gjkf0d.gif) fiogf49gjkf0d
fiogf49gjkf0d.gif) fiogf49gjkf0dFigura 3. Cuantificación manual mediante la función “Manual Tag” del software Image-Pro Plus 5.0®.">
fiogf49gjkf0dFigura 3. Cuantificación manual mediante la función “Manual Tag” del software Image-Pro Plus 5.0®.">
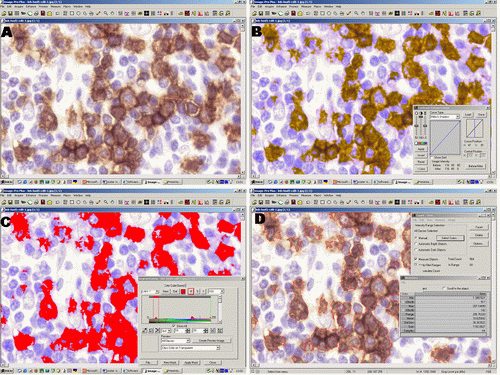 fiogf49gjkf0dFigura 4. Proceso de cuantificación semiautomática. (A) área representativa escaneada a 40X, (B, C, D) recuento del número de células positivas tras aplicación de la macro específica por patrón de tinción.">
fiogf49gjkf0dFigura 4. Proceso de cuantificación semiautomática. (A) área representativa escaneada a 40X, (B, C, D) recuento del número de células positivas tras aplicación de la macro específica por patrón de tinción.">
 fiogf49gjkf0d
fiogf49gjkf0d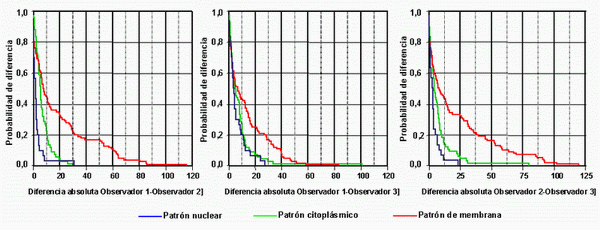 fiogf49gjkf0d
fiogf49gjkf0d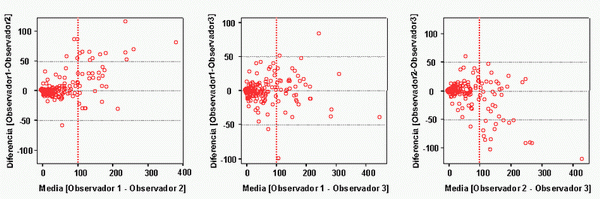 fiogf49gjkf0dFigura 7. Representación de Bland-Altman. Diferencias en las mediciones de diferentes observadores en relación con el promedio de las mediciones. Se puede observar un aumento de las diferencias cuando el numero de células teñidas es superior a 100 células/campo.">
fiogf49gjkf0dFigura 7. Representación de Bland-Altman. Diferencias en las mediciones de diferentes observadores en relación con el promedio de las mediciones. Se puede observar un aumento de las diferencias cuando el numero de células teñidas es superior a 100 células/campo.">
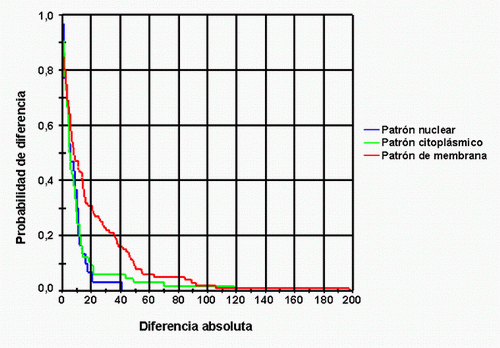 fiogf49gjkf0dFigura 8. Curvas de Kaplan-Meier (análisis entre Image-Pro Plus y los observadores): Probabilidad de obtener diferencias en valor absoluto entre la medición semi-automatica y la media de los tres observadores para tinciones con patrón nuclear, citoplásmico y de membrana.">
fiogf49gjkf0dFigura 8. Curvas de Kaplan-Meier (análisis entre Image-Pro Plus y los observadores): Probabilidad de obtener diferencias en valor absoluto entre la medición semi-automatica y la media de los tres observadores para tinciones con patrón nuclear, citoplásmico y de membrana.">
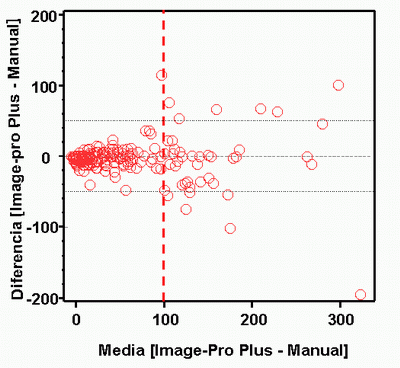 fiogf49gjkf0dFigura 9. Representación de Bland-Altman. Diferencias en la medición realizada con el software Image-pro Plus y la media de las mediciones realizadas por parte de los tres observadores en relación con el promedio de las mediciones. Se puede observar un aumento de las diferencias cuando el numero de células teñidas es superior a 100 células/campo.">
fiogf49gjkf0dFigura 9. Representación de Bland-Altman. Diferencias en la medición realizada con el software Image-pro Plus y la media de las mediciones realizadas por parte de los tres observadores en relación con el promedio de las mediciones. Se puede observar un aumento de las diferencias cuando el numero de células teñidas es superior a 100 células/campo.">