

|
Patrón de Metilación génica en el cáncer de la vesícula biliar Juan Carlos Roa S*, Angélica Melo A*, Leonardo Anabalón*, Oscar Tapia E*, Xabier de Aretxabala U**, Iván Roa E*** |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
La metilación de la región promotora de genes es un importante mecanismo en la inactivación de genes supresores de tumores.
Objetivo Analizar el patrón metilación de importantes genes involucrados en procesos carcinogenéticos de la vesícula biliar, correlacionándolas con su expresión inmunohistoquímica, características anatomoclínicas y sobrevida de los pacientes.
Material y Método Se seleccionaron 20 casos de cáncer de vesícula biliar del banco de tumores congelados. El ADN extraído fue analizado mediante el test de metilación específica por PCR para los Genes CDKN2A (p16), hMLH1, APC, FHIT y CDH1 (Caderina –E). Se obtuvo datos anatomoclínicos y seguimiento de la totalidad de los pacientes.
Resultados: La totalidad de los casos correspondió a tumores avanzados (95%) histológicamente moderada o pobremente diferenciados (95%).Se observó metilación del área promotora de los genes en 5%, 20%, 30%, 40%, 65% y un patrón inmunohistoquímico alterado (PIA) en 5%, 35%, 21% ,25%, 66% para los genes hMLH1, CDKN2A, FHIT, APC y Caderina-E respectivamente. El indice de concordancia Kappa fue casi perfecto entre metilación del área promotora y PIA para los genes hMLH1 y Caderina E y substancial para APC. No se encontró correlación entre sobreviva y la metilación de los genes estudiados.
Conclusiones: La alta frecuencia de de metilación génica (con la excepción de hMLH1) y la alta correlación de PIA y metilación del área promotora génica para los genes hMLH1, APC y Caderina E, sugieren que la metilación es un mecanismo importante en su inactivacion, jugando probablemente un rol en la tumorogénesis vesicular.
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
Existen escasos estudios comunicados respecto de la carcinogénesis vesicular se han enfocado a detectar la mutación de genes dominantes como K-ras (1-4)o la inactivación de genes supresores de tumores mediante Mutación o deleción alélica(2, 5-8). Recientemente estudios de extensa alelotipificación cromosómica han mostrado que existen deleciones alélicas múltiples en diferentes sitios del genoma humano, sugiriendo la participación de numerosos genes supresores de tumores en esta neoplasia(9).
La metilación del ADN es el cambio epigenético más frecuente e importante en tumores humanos, incidiendo directamente en la inactivación de los genes supresores de tumores como mecanismo alterno a la mutación y deleción alélica, La inactivación por metilación afecta directamente procesos tan importantes como el ciclo celular, la reparación del ADN y aquellos involucrados en la invasión y metástasis. (10, 11)
En las células tumorales existe un estado de hipometilación del ADN genómico p, sin embargo, se observa hipermetilación en regiones ricas en citocinas y guaninas (islotes CpG), en las zonas que están ubicadas frecuentemente cerca o dentro de las áreas promotoras génicas, en aproximadamente la mitad de los genes humanos conocidos. (12-14). La hipermetilación de estas áreas produce represión transcripcional debido a un cambio en la estructura de la cromatina, que la hace inaccesible a los factores de transcripción, y de esta manera inactiva a los genes involucrados en las distintas vías carcinogenéticas. Este fenómeno ha sido reportado en varios tipos de tumores, lo que ha permitido establecer patrones específicos de metilación en diferentes subgrupos de tumores. (15, 16). El adecuado entendimiento de los perfiles de metilación tumoral puede tener un impacto en importantes problemas clínicos como la detección precoz, quimioprevención, pronóstico y tratamiento de las enfermedades neoplásicas (10)
Nuestro objetivo fue examinar el patrón de metilación de la región promotora de genes involucrados en distintas vías carcinogenéticas en carcinomas vesiculares avanzados, además relacionar su expresión inmunohistoquímica y algunas características clínicas y morfológicas asociadas a pronósticos diferentes.
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
Casos: se seleccionaron 20 casos de pacientes con cancer avanzado de vesícula biliar del banco de tumores del Departamento de Anatomía Patológica de la Universidad de la Frontera. Todas las muestras fueron mantenidas en gel criopreservante (Jung, Alemania) a -70ºC. Se seleccionó un área tumoral mediante corte de congelación y se fragmentó en cámara fría. En todos los pacientes se obtuvo seguimiento completo y las características clínicas de la serie se obtuvieron desde los registros médicos. Para la clasificación morfológica de los tumores se utilizaron los criterios de la OMS(17) y la clasificación TNM(18). Se seleccionó los genes supresores de tumores relacionados con el ciclo celular FHIT (Fragile Histidine Triad), CDKN2A (p16) y APC (Adenomatous Poliposis Coli). El gen hMLH1 (Human Mut Homologue 1) (19)probablemente el más importante de los genes reparadores de ADN genómico, relacionado con inestabilidad microsatelital (20)y el gen CDH1 (Caderina E), que codifica una proteína de transmembrana que participa en el complejo de moléculas de adhesión involucrada en la metástasis e invasión tumoral. (21) El criterio de selección de los genes estudiados, se basó básicamente en la incidencia de estos en las distintas vías carcinogenéticas de tumores digestivos en humanos. (22, 23) Extracción del ADN: Se realizó según protocolo del Kit de Aislamiento de ADN Genómico Puregene (GENTRA, USA). Brevemente, los fragmentos de tejido se incubaron a 65ºC en tubos Eppendorf de 1,5ml con solución de lisis celular y proteinasa K (10mg/ml) hasta observar lisis tisular total. Las proteínas fueron eliminadas por precipitación en acetato de amonio 7,5M pH 7,5. El ADN fue precipitado con isopropanol, lavado con etanol 70%, resuspendido en solución de hidratación (buffer TE pH 8.0) y almacenado a -20ºC. Prueba de metilación especifica mediante PCR (MSP) Modificación con Bisulfito: El principio de la modificación de ADN con la técnica de bisulfito de sodio se basa en su capacidad de convertir a todos los residuos de citosina (C) no metiladas en uracilos (U) mediante deaminación, la citosina metilada es resistente a la reacción y permanece como citosina(24). Los iniciadores de PCR utilizados aprovechan estas diferencias para discriminar entre aquellas secuencias metiladas y no metiladas. Se usó protocolo previamente descrito. (23, 24). Amplificación del ADN: Las secuencias de partidores y condiciones de PCR han sido previamente descritas. (23) Las reacciones fueron realizadas con aproximadamente 100 ng de ADN genómico modificado, 0,2 µM de cada iniciador, 200 µM de cada uno de los desoxinucleótidos trifosfatos, 1,5 mM de MgCl y 0,75 U de Taq Polimerasa (Promega) en un volumen final de 25 µl. Se usó como control negativo 100 ng de ADN genómico sin modificar. Como control positivo de metilación se utilizó ADN genómico comercial (Promega) metilado con SssI (Biolab) y como control negativo, ADN genómico (Promega) no metilado modificado. Los productos de PCR, fueron visualizados mediante electroforesis en geles de poliacrilamida al 12% en buffer TBE y de agarosa al 2% en buffer TAE, teñidos con bromuro de etidio (Figura 1). Se utilizó el índice de metilación como un indicador de la proporción de regiones promotoras metiladas respecto de la totalidad de los genes estudiados y se calcula dividiendo el número de genes metilados por el número de genes analizados. Estudio Inmunohistoquímico: Se utilizó la técnica estándar Streptavidina-Biotina (Biogenex) para tejidos frescos congelados. No se realizó recuperación antigénica La incubación de los anticuerpos primarios APC (Clon C-20), p16 (clon F-12), E-Cadherin (clon C-19) de Sta. Cruz California. Y FHIT policlonal, MLH1 (Clon 14) de Zymed laboratories, Inc South San Francisco, California) en concentración 1:50. Posteriormente fueron incubados con anticuerpo secundario 1:320 (multilink biotinylated) (Biogenex), durante una hora y finalmente revelados con diaminobenzidina. En cada prueba se emplearon controles negativos y positivos. En los controles negativos se sustituyó el anticuerpo primario por suero normal, y como control positivo se utilizaron casos de carcinomas del colon, en los que previamente se había demostrado positividad para estos antígenos. Patrón inmunohistoquímico alterado (PIA). Se consideró la presencia de un PIA cuando se obtuvo ausencia de tinción inmunohistoquímica en el tumor y positividad en el infiltrado inflamatorio o en la mucosa tumoral adyacente. Estos genes supresores normalmente producen una proteína que ejercen el efecto supresor tumoral sin embargo al producirse la inactivacion del gen, sea esta por metilación, mutación, deleción o combinación de ellos, no produce la proteína y por lo tanto no es detectada mediante la IHQ (Figura 2). Análisis Estadístico. La relación entre el estado de metilación génica con variables categóricas tales como sexo, raza, tipo histológico usando la prueba de X2 o la prueba exacta de Fisher. Para variables numéricas como edad se utilizó la prueba de t-student.. Para el calculo de sobrevida y su asociación con parámetros morfológicos y clínicos y metilación génica se construyeron curvas de Kaplan Meier y fueron comparadas utilizando la prueba de log-rank y el modelo de Cox Hazard. Para la medición de la concordancia entre metilación y PIA se utilizó la prueba estadísticoa de Kappa. En este, el valor Kappa de 1.0 fue considerado perfecto los valores entre 1.0 y 0.75 fueron considerados casi perfectos, entre 0.45 y 0.75 substanciales, entre 0.20 y 0.45 moderados y menor a 0.20 malo. (ref Muñoz)
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
Características clínicas
Las características generales del grupo estudiado se observan en la Tabla 1. Este fue mayoritariamente de sexo femenino (80%) y no mapuche (85%). La serie presentó una edad promedio de 62.5 años. La sobrevida en promedio fue de 10,7 meses con un rango de 1 a 66 meses.
La mayoría de los casos se correspondió a tumores subserosos o serosos (95%). En relación al grado de diferenciación, la mayoría correspondió a tumores poco y moderadamente diferenciados (55% y 40% respectivamente). La totalidad de los casos correspondieron a adenocarcinomas tubulares.
Estado de metilación génica.
Las frecuencias de metilación de la región promotora de los genes estudiados en los 20 casos de tumores de vesícula biliar analizados (Tabla 2), fluctuaron entre 5% para hMLH1 y 65% para CDH1. Los genes p16, FHIT y APC presentaron frecuencias de metilación similares (20% , 30% y 40% respectivamente). Observamos una interesante tendencia entre la metilación de CDH1 y la presencia de tumores poco diferenciados (p=0.06).
Estudio Inmunohistoquímico: Encontramos un pia en 5%, 35%, 21% ,25%, 66% para los genes hMLH1, CDKN2A, FHIT, APC y Caderina-E respectivamente. La concordancia entre metilación del área promotora y PIA presento valores elevados de Kappa (K) para los genes hMLH1 (K=1.0; p<0.0001), APC (K=0.66, p<0.001) y Caderina-E (K=0.75; p<0.001). Par el gen CDKN2A el porcentaje de acuerdo fue de 59% con nivel 0.39 de Kappa, sin embargo se observo un p<0.05. No se observó correlación entre el PIA y sobrevida ni características morfológicas de los tumores.
No se encontró relación entre la sobrevida de los pacientes con el patrón de metilación de los genes analizados.
El índice de metilación se resume en la Tabla 3. El 75% de los casos presentaron 1 o más genes metilados es decir un índice de metilación de 0.2 o superior.
Tabla 1. Características generales y morfológicas del grupo estudiado(n=20)
Tabla 2. Metilación del promotor y Patrón IHQ alterado.
Tabla 3. Indice de metilación.
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
Trabajo financiado por Fondecyt 1050603 y Diufro 120333.
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
1 - Roa JC, Roa I, de Aretxabala X, Melo A, Faria G, Tapia O. [K-ras gene mutation in gallbladder carcinoma]. Rev Med Chil 2004;132:955-60.
2 - Wistuba, II, Miquel JF, Gazdar AF, Albores-Saavedra J. Gallbladder adenomas have molecular abnormalities different from those present in gallbladder carcinomas. Hum Pathol 1999;30:21-5. 3 - Rashid A. Cellular and molecular biology of biliary tract cancers. Surg Oncol Clin N Am 2002;11:995-1009. 4 - Ajiki T, Fujimori T, Onoyama H, Yamamoto M, Kitazawa S, Maeda S, et al. K-ras gene mutation in gall bladder carcinomas and dysplasia. Gut 1996;38:426-9. 5 - Wistuba, II, Sugio K, Hung J, Kishimoto Y, Virmani AK, Roa I, et al. Allele-specific mutations involved in the pathogenesis of endemic gallbladder carcinoma in Chile. Cancer Res 1995;55:2511-5. 6 - Kim YT, Kim J, Jang YH, Lee WJ, Ryu JK, Park YK, et al. Genetic alterations in gallbladder adenoma, dysplasia and carcinoma. Cancer Lett 2001;169:59-68. 7 - Nakayama K, Konno M, Kanzaki A, Morikawa T, Miyashita H, Fujioka T, et al. Allelotype analysis of gallbladder carcinoma associated with anomalous junction of pancreaticobiliary duct. Cancer Lett 2001;166:135-41. 8 - Hidaka E, Yanagisawa A, Sakai Y, Seki M, Kitagawa T, Setoguchi T, et al. Losses of heterozygosity on chromosomes 17p and 9p/18q may play important roles in early and advanced phases of gallbladder carcinogenesis. J Cancer Res Clin Oncol 1999;125:439-43. 9 - Wistuba, II, Tang M, Maitra A, Alvarez H, Troncoso P, Pimentel F, et al. Genome-wide allelotyping analysis reveals multiple sites of allelic loss in gallbladder carcinoma. Cancer Res 2001;61:3795-800. 10 - Herman JG, Baylin SB. Gene silencing in cancer in association with promoter hypermethylation. N Engl J Med 2003;349:2042-54. 11 - Costello JF, Fruhwald MC, Smiraglia DJ, Rush LJ, Robertson GP, Gao X, et al. Aberrant CpG-island methylation has non-random and tumour-type-specific patterns. Nat Genet 2000;24:132-8. 12 - Jones PA, Laird PW. Cancer epigenetics comes of age. Nat Genet 1999;21:163-7. 13 - Wolffe AP, Matzke MA. Epigenetics: regulation through repression. Science 1999;286:481-6. 14 - Takai D, Jones PA. Comprehensive analysis of CpG islands in human chromosomes 21 and 22. Proc Natl Acad Sci U S A 2002;99:3740-5. Epub 2002 Mar 12. 15 - Robertson KD, Jones PA. DNA methylation: past, present and future directions. Carcinogenesis 2000;21:461-7. 16 - Merlo A, Herman JG, Mao L, Lee DJ, Gabrielson E, Burger PC, et al. 5' CpG island methylation is associated with transcriptional silencing of the tumour suppressor p16/CDKN2/MTS1 in human cancers. Nat Med 1995;1:686-92. 17 - Stanley Hamilton LA. Tumours of the Digestive System. Lyon: IARC Press; 2000. 18 - The new TNM classification in gastroenterology (1997). Endoscopy 1998;30:643-9. 19 - Christoph. CDL. Tumor Genome Instability. In: Kenneth. VBK, editor. The genetic basis of cancer. New York: Mc Graw Hill; 2002. p. 129-130. 20 - Roa JC, Roa I, Correa P, Vo Q, Araya JC, Villaseca M, et al. Microsatellite instability in preneoplastic and neoplastic lesions of the gallbladder. J Gastroenterol 2005;40:79-86. 21 - Wajed SA, Laird PW, DeMeester TR. DNA methylation: an alternative pathway to cancer. Ann Surg 2001;234:10-20. 22 - Berx G, Van Roy F. The E-cadherin/catenin complex: an important gatekeeper in breast cancer tumorigenesis and malignant progression. Breast Cancer Res 2001;3:289-93. Epub 2001 Jun 28. 23 - Roa JC, Anabalon L, Tapia O, Martinez J, Araya JC, Villaseca M, et al. [Promoter methylation profile in breast cancer]. Rev Med Chil 2004;132:1069-77. 24 - Herman JG, Graff JR, Myohanen S, Nelkin BD, Baylin SB. Methylation-specific PCR: a novel PCR assay for methylation status of CpG islands. Proc Natl Acad Sci U S A 1996;93:9821-6. 25 - Takahashi T, Shivapurkar N, Reddy J, Shigematsu H, Miyajima K, Suzuki M, et al. DNA methylation profiles of lymphoid and hematopoietic malignancies. Clin Cancer Res 2004;10:2928-35. 26 - Paz MF, Fraga MF, Avila S, Guo M, Pollan M, Herman JG, et al. A systematic profile of DNA methylation in human cancer cell lines. Cancer Res 2003;63:1114-21. 27 - Yang B, House MG, Guo M, Herman JG, Clark DP. Promoter methylation profiles of tumor suppressor genes in intrahepatic and extrahepatic cholangiocarcinoma. Mod Pathol 2004;1:1. 28 - Aaltonen LA, Peltomaki P, Leach FS, Sistonen P, Pylkkanen L, Mecklin JP, et al. Clues to the pathogenesis of familial colorectal cancer. Science 1993;260:812-6. 29 - Mitchell RJ, Farrington SM, Dunlop MG, Campbell H. Mismatch repair genes hMLH1 and hMSH2 and colorectal cancer: a HuGE review. Am J Epidemiol 2002;156:885-902. 30 - Fedier A, Fink D. Mutations in DNA mismatch repair genes: implications for DNA damage signaling and drug sensitivity (review). Int J Oncol 2004;24:1039-47. 31 - Kohya N, Miyazaki K, Matsukura S, Yakushiji H, Kitajima Y, Kitahara K, et al. Deficient expression of O(6)-methylguanine-DNA methyltransferase combined with mismatch-repair proteins hMLH1 and hMSH2 is related to poor prognosis in human biliary tract carcinoma. Ann Surg Oncol 2002;9:371-9. 32 - Takahashi T, Shivapurkar N, Riquelme E, Shigematsu H, Reddy J, Suzuki M, et al. Aberrant promoter hypermethylation of multiple genes in gallbladder carcinoma and chronic cholecystitis. Clin Cancer Res 2004;10:6126-33. 33 - House MG, Wistuba, II, Argani P, Guo M, Schulick RD, Hruban RH, et al. Progression of gene hypermethylation in gallstone disease leading to gallbladder cancer. Ann Surg Oncol 2003;10:882-9. 34 - Lee JH, Park SJ, Abraham SC, Seo JS, Nam JH, Choi C, et al. Frequent CpG island methylation in precursor lesions and early gastric adenocarcinomas. Oncogene 2004;23:4646-54. 35 - Ueki T, Hsing AW, Gao YT, Wang BS, Shen MC, Cheng J, et al. Alterations of p16 and prognosis in biliary tract cancers from a population-based study in China. Clin Cancer Res 2004;10:1717-25. 36 - Shi YZ, Hui AM, Li X, Takayama T, Makuuchi M. Overexpression of retinoblastoma protein predicts decreased survival and correlates with loss of p16INK4 protein in gallbladder carcinomas. Clin Cancer Res 2000;6:4096-100. 37 - Koda M, Yashima K, Kawaguchi K, Andachi H, Hosoda A, Shiota G, et al. Expression of Fhit, Mlh1, and P53 protein in human gallbladder carcinoma. Cancer Lett 2003;199:131-8. 38 - Wistuba, II, Ashfaq R, Maitra A, Alvarez H, Riquelme E, Gazdar AF. Fragile histidine triad gene abnormalities in the pathogenesis of gallbladder carcinoma. Am J Pathol 2002;160:2073-9. 39 - Roa I, Ibacache G, Melo A, Morales E, Villaseca M, Araya J, et al. [Subserous gallbladder carcinoma: expression of cadherine-catenine complex]. Rev Med Chil 2002;130:1349-57. 40 - Tozawa T, Tamura G, Honda T, Nawata S, Kimura W, Makino N, et al. Promoter hypermethylation of DAP-kinase is associated with poor survival in primary biliary tract carcinoma patients. Cancer Sci 2004;95:736-40.
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
- Cesáreo Corbacho Cuevas (25/10/2005 20:04:10)
- Cesáreo Corbacho Cuevas (26/10/2005 10:10:30)
- Juliana Fariña (30/10/2005 2:36:03)
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
|
Web mantenido y actualizado por el Servicio de informática uclm. Modificado: 16/06/2015 14:10:50
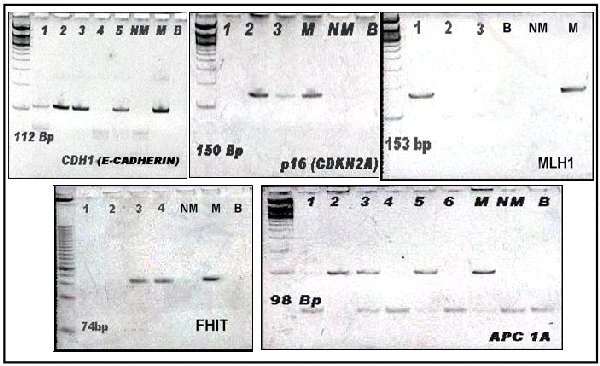
.jpg) fiogf49gjkf0dFigura 1. Fotografía compuesta de geles de agarosa que muestra el resultado del análisis de metilación en los 5 genes estudiados donde se puede observar el tamaño variable de las bandas amplificadas.
M= control positivo ADN genómico metilado y modificado, NM= control negativo ADN genómico no metilado y modificado y B= Blanco.
">
fiogf49gjkf0dFigura 1. Fotografía compuesta de geles de agarosa que muestra el resultado del análisis de metilación en los 5 genes estudiados donde se puede observar el tamaño variable de las bandas amplificadas.
M= control positivo ADN genómico metilado y modificado, NM= control negativo ADN genómico no metilado y modificado y B= Blanco.
">
 fiogf49gjkf0dFigura 2. Ejemplos de patrones de inmunohistoquímicos observados para los genes estudiados. Nótese la negatividad de las células tumorales para los genes hMLH1 (D) y CDKN2A(p16)(E) en relación con las células inflamatorias adyacentes (control interno positivo) y las áreas tumorales reactivas y no reactivas para CDH1 (B).
">
fiogf49gjkf0dFigura 2. Ejemplos de patrones de inmunohistoquímicos observados para los genes estudiados. Nótese la negatividad de las células tumorales para los genes hMLH1 (D) y CDKN2A(p16)(E) en relación con las células inflamatorias adyacentes (control interno positivo) y las áreas tumorales reactivas y no reactivas para CDH1 (B).
">