|
|
p16/INK4 y p14/ARF en tumores primitivos neuroectodérmicos (PNETs) del sistema nervioso central
MM Inda
,
T Tuñón
,
JM Martínez-Peñuela
y
JS Castresana
Departamento de Genética
Universidad de Navarra
Irunlarrea 1 Pamplona
31008
Navarra
España
|
|
|
|
|
|
|
|
Resumen
|
|
|
|
INTRODUCCIÓN y OBJETIVOS
Los PNETs supratentoriales (sPNETs) son similares morfológicamente a los PNETs infratentoriales o meduloblastomas (MBs), pero muestran diferente comportamiento biológico, siendo, los sPNETs más agresivos que los MBs.
Ante la sospecha de que estos tumores presenten diferencias genéticas específicas de grupo, que puedan ayudar a elegir un tratamiento más específico, se ha estudiado el patrón de alteración de los genes p16/INK4 y p14/ARF en tumores primarios.
MATERIAL Y MÉTODOS
Se extrajo el DNA de 31 tumores incluidos en parafina (8 sPNETs y 23 MBs), y se estudió la presencia de deleciones homocigóticas (HD) del locus CDKN2A (p16/INK4, exón 1-alfa; y p14/ARF, exón 1-beta) en 9p21 mediante PCR diferencial.
También se estudió mediante MSP (Methylation Specific PCR) el estado de metilación de los promotores de p16/INK4 y p14/ARF.
Se utilizó el test exacto de Fisher para comparar los resultados obtenidos.
RESULTADOS
Ninguna de las muestras analizadas presentó deleción homocigótica ni de p16/INK4 (0/4 sPNETs y 0/18 MBs) ni de p14/ARF (0/8 sPNETs y 0/22 MBs).
Se encontró metilación del promotor de p16/INK4 en 1/8 sPNETs (13%) y en 1/23 MBs (4%). Se encontró hipermetilación del promotor de p14/ARF en 5/8 sPNETs (63%) y en 3/20 MBs (15%).
Se encontraron diferencias significativas (p<0.05) en el patrón de metilación de p14/ARF.
CONCLUSIÓN
p16/INK4 no se muestra frecuentemente alterado en MBs/PNETs ni por pérdida homocigótica ni por hipermetilación de su promotor. Estos resultados concuerdan con lo encontrado por otros autores.
Hemos demostrado una frecuencia superior de hipermetilación del promotor de p14/ARF en MBs (15%) a la hallada en otros estudios. La frecuencia de metilación de dicho promotor es significativamente (p<0.05) superior en los sPNETs. Ello hace pensar que p14/ARF pueda jugar un importante papel en la génesis de los sPNETs, marcando así una diferencia genotípica frente a los MBs, morfológicamente iguales.
|
|
|
|
|
|
|
Introducción
|
|
|
|
Los tumores del sistema nervioso central (SNC) constituyen el grupo más frecuente de tumores sólidos pediátricos. Dentro de los tumores del SNC, se distinguen los tumores primitivos neuroectodérmicos (PNETs) que son los tumores malignos (grado IV según la OMS) más frecuentes en niños. Los meduloblastomas (MBs), son PNETs infratentoriales localizados en el cerebelo y constituyen el 65% de los tumores intracraneales en niños menores de 5 años.
Los PNETs supratentoriales (sPNETs) son similares morfológicamente a los MBs, pero muestran diferente comportamiento biológico, siendo los sPNETs más agresivos que los MBs.
Ante la sospecha de que estos tumores presenten diferencias genéticas específicas de grupo, que puedan ayudar a elegir un tratamiento más específico, se ha estudiado el patrón de alteración de los genes p16/INK4 y p14/ARF en tumores primarios.
|
|
|
|
|
|
|
Material y Métodos
|
|
|
|
Se extrajo el DNA de 31 muestras de MB/sPNET incluidas en parafina (8 sPNETs y 23 MBs) mediante métodos convencionales y se estudió la presencia de deleciones homocigóticas (HD) del locus CDKN2A (p16/INK4, exón 1-alfa, y p14/ARF, exón 1-beta) en 9p21 mediante PCR diferencial.
También se estudió mediante MSP (Methylation Specific PCR) el estado de metilación de los promotores de p16/INK4 y p14/ARF. Un microgramo de DNA fue modificado mediante bisulfito según Herman et al. y, posteriormente, fue amplificado con cebadores específicos de la secuencia metilada y no metilada.
Se utilizó el test exacto de Fisher para comparar los resultados obtenidos.
|
|
|
|
|
|
|
Resultados
|
|
|
|
Los resultados obtenidos tanto para el estudio de deleciones homocigóticas como de hipermetilación del promotor de p16/INK4 y p14/ARF, se encuentran resumidos en las tablas 1 y 2.
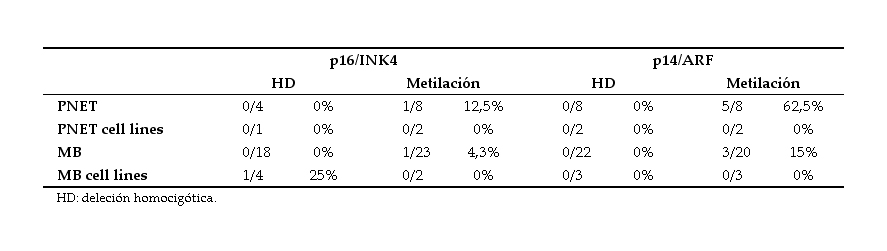
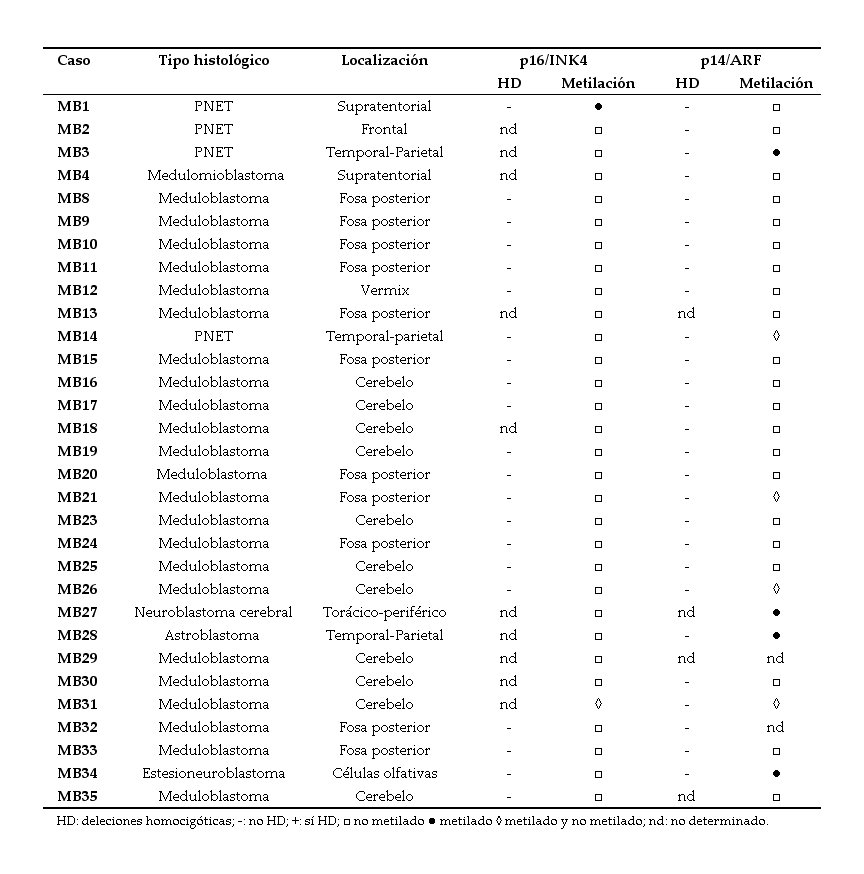
Ninguna de las muestras analizadas presentó deleción homocigótica ni de p16/INK4 (0/4 sPNETs y 0/18 MBs) ni de p14/ARF (0/8 sPNETs y 0/22 MBs) (figuras 1 y 2).
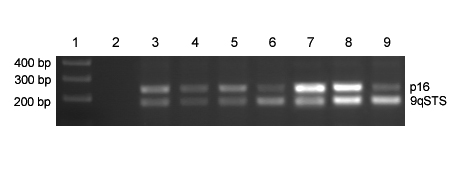
Se encontró metilación del promotor de p16/INK4 en 1/8 sPNETs (13%) y en 1/23 MBs (4%) (figura 3). Se encontró hipermetilación del promotor de p14/ARF en 5/8 sPNETs (63%) y en 3/20 MBs (15%) (figura 4).
Se encontraron diferencias significativas (p<0.05) en el patrón de metilación de p14/ARF.
|
 Zoom Zoom
|
|
|
|
 Zoom Zoom
|
|
|
|
 Zoom Zoom
|
|
|
Deleciones homocigóticas (HD) de p16/INK4 en MB/sPNET primarios. Se amplificó un fragmento del gen p16/INK4 (exón 1-alfa) de 235 pb (banda superior) junto a una secuencia control 9qSTS (179 pb, banda inferior). Los productos de PCR fueron separados en un gel de agarosa al 2.5% teñido con bromuro de etidio. DNA de sangre periférica de donantes sanos fue usado como control negativo de HD de p16/INK4 (calles 3 y 4). Calle 1: Marcador de peso molecular, 1 Kb plus-ladder; calle 2: control negativo de la PCR; calles 3 y 4: controles negativos de HD de p16/INK4; calles 5-9: muestras de MB y sPNET. Se considera que existe una HD cuando el cociente de intensidades entre la banda superior y la inferior es menor a un tercio del cociente calculado para los controles negativos. No se encontró ninguna HD de p16/INK4 en ninguna de las muestras analizadas.
|
 Zoom Zoom
|
|
|
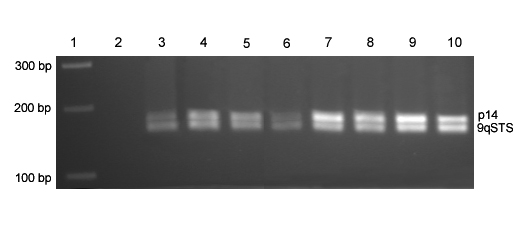
Deleciones homocigóticas (HD) de p14/ARF en MB/sPNET primarios. Se amplificó un fragmento del gen p14/ARF (exón 1-beta) de 174 pb (banda superior) junto a una secuencia control 9qSTS (179 pb, banda inferior). Los productos de PCR fueron separados en un gel de agarosa al 2.5% teñido con bromuro de etidio. DNA de sangre periférica de donantes sanos fue usado como control negativo de HD de p14/ARF (calles 3 y 4). Calle 1: Marcador de peso molecular, 1 Kb plus-ladder; calle 2: control negativo de la PCR; calles 3 y 4: controles negativos de HD de p14/ARF; calles 5-10: muestras de MB y sPNET. Se considera que existe una HD cuando el cociente de intensidades entre la banda superior y la inferior es menor a un tercio del cociente calculado para los controles negativos. No se encontró ninguna HD de p14/ARF en ninguna de las muestras analizadas.
|
 Zoom Zoom
|
|
|
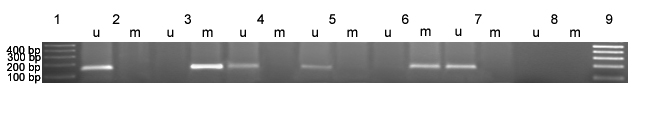
Hipermetilación del promotor de p16/INK4. Se usó la técnica de MSP para detectar hipermetilación del promotor de p16/INK4. DNA de sangre periférica de donantes sanos modificado fue usado como control negativo de metilación (2). El DNA de la línea celular T24 fue usado como control positivo de metilación del promotor de p16/INK4 (3). Los productos de PCR fueron visualizados en un gel de agarosa al 2% teñido con bromuro de etidio tras electroforesis. U: corresponde al producto de la PCR realizada con los cebadores diseñados para amplificar la secuencia no metilada (151 pb). M: corresponde al producto de la PCR realizada con los cebadores diseñados para amplificar la secuencia metilada (150 pb). 1 y 9: Marcador de peso molecular, 1 Kb plus-ladder; 2: control negativo de metilación; 3: control positivo de metilación; 4-7: muestras de MB/sPNET; 8: control negativo de la PCR. Se encontró que MB1 (calle 6) y MB16 (calle 7) presentaban hipermetilación del promotor de p16/INK4. Las muestras MB12 y MB15 (calles 4 y 5) no presentaban metilación de dicho promotor. En la muestra MB16, se observó una banda tanto en (U) como en (M).
|
 Zoom Zoom
|
|
|
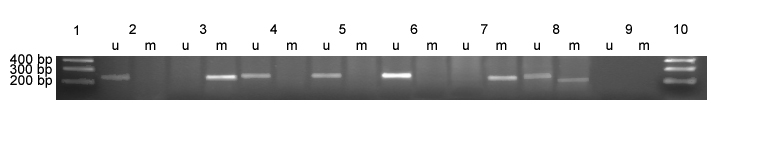
Hipermetilación del promotor de p14/ARF. Se usó la técnica de MSP para detectar hipermetilación del promotor de p14/ARF. DNA de sangre periférica de donantes sanos modificado fue usado como control negativo de metilación (2). Como control positivo de metilación del promotor de p14/ARF se usó CpGTM Universal methylated DNA (3). Los productos de PCR fueron visualizados en un gel de agarosa al 2% teñido con bromuro de etidio tras electroforesis. U: corresponde al producto de la PCR realizada con los cebadores diseñados para amplificar la secuencia no metilada (132 pb). M: corresponde al producto de la PCR realizada con los cebadores diseñados para amplificar la secuencia metilada (122 pb). 1 y 10: Marcador de peso molecular, 1 Kb plus-ladder; 2: control negativo de metilación; 3: control positivo de metilación; 4-8: muestras de MB/sPNET; 9: control negativo de la PCR. Se encontró que MB34 (calle 7) y MB26 (calle 8) presentaban hipermetilación del promotor de p14/ARF. Las muestras MB19, MB20 y MB27 (calles 4-6) no presentaban metilación de dicho promotor. En la muestra MB26, se observó una banda tanto en (U) como en (M).
|
|
|
|
|
|
|
|
Discusión
|
|
|
|
p16/INK4 no se encuentra frecuentemente alterado en MBs/PNETs ni por pérdida homocigótica ni por hipermetilación de su promotor. Estos resultados concuerdan con lo encontrado por otros autores.
En el presente estudio, se ha encontrado una frecuencia superior de hipermetilación del promotor de p14/ARF en MBs (15%) de la hallada en otros estudios (Lindsey et al., 2003) (7%). La frecuencia de metilación de dicho promotor es significativamente (p<0.05) superior en los sPNETs que en los MBs. Ello hace pensar en que p14/ARF puede jugar un importante papel en la génesis de sPNETs, marcando así una diferencia genotípica frente a los MBs, morfológicamente iguales. Estos estudios deberían ser corroborados con estudios en series mayores de tumores.
|
|
|
|
|
|
|
Bibliografia
|
|
|
|
1. Barker, F.G., et al., P16 deletion and mutation analysis in human brain tumors. J Neurooncol, 1997. 31(1-2): p. 17-23.
2. Fruhwald, M.C., et al., Aberrant promoter methylation of previously unidentified target genes is a common abnormality in medulloblastomas-implications for tumor biology and potential clinical utility. Oncogene, 2001. 20(36): p. 5033-42.
3. Harada, K., et al., Aberrant promoter methylation and silencing of the RASSF1A gene in pediatric tumors and cell lines. Oncogene, 2002. 21(27): p. 4345-9.
4. Jen, J., et al., Deletion of p16 and p15 genes in brain tumors. Cancer Res, 1994. 54(24): p. 6353-8.
5. Lindsey, J.C., et al., Identification of tumour-specific epigenetic events in medulloblastoma development by hypermethylation profiling. Carcinogenesis, 2003 (E. publication; ahead of print).
6. Raffel, C., et al., The multiple tumor suppressor 1/cyclin-dependent kinase inhibitor 2 gene in human central nervous system primitive neuroectodermal tumor. Neurosurgery, 1995. 36(5): p. 971-4; discussion 974-5.
7. Sato, K., et al., Infrequent alterations of the p15, p16, CDK4 and cyclin D1 genes in non-astrocytic human brain tumors. Int J Cancer, 1996. 66(3): p. 305-8.
|
|
|
|
|
|
|
NOTA:
Esto es un foro médico profesional, que no tiene como objetivo ofrecer consejo médico o de salud. Los mensajes enviados a este foro solicitando estos consejos, no serán atendidos. La información científica ofrecida está refrendada por las referencias y bibliografía correspondientes y de su veracidad son responsables sus autores. La participación en este Congreso es gratuita.
Todos los derechos reservados (C) UNINET 2003
Versión 1.5
Si experimenta cualquier problema en la edición de su trabajo envie un mensaje a la siguiente dirección: info.conganat@infomed.sld.cu